
图片来源:doi:10.3389/fpls.2015.00189
摘要:多倍体无参植物RNA-seq经典文章解读
油茶,又名茶子树、茶油树,因其种子可榨油,被列入世界四大木本油料树种之一。油茶主要生长在我国南方高山及丘陵地带,作为一种常绿阔叶乔木,油茶具有一定的冷适应性。最近发表的一篇《BMC Genomics》文章,就利用了RNA-seq技术,探讨了这种冷适应性可能的分子机制。
英文题目:Leaf transcriptome analysis of a subtropicalevergreen broadleaf plant, wild oil-teacamellia (Camellia oleifera), revealingcandidate genes for cold acclimation
中文题目:对亚热带常绿阔叶植物野生油茶(Camellia oleifera)的叶片转录组分析揭示冷适应性相关候选基因
期刊名:BMC Genomics发表时间: 2017年影响因子:3.867
研究背景:冷耐受性是在地理分布范围上影响植物种类和作物产量的关键因素之一。冷适应可以在未冰冻的低温阶段起到增强植物的冷耐受性的作用。作为一种常绿阔叶植物,油茶表现出相对较强的耐受寒冷低温的能力。
研究目的:通过对不同纬度和海拔油茶转录组变化的分析,发现冷适应相关基因。
材料方法:
研究地点及采样方法情况
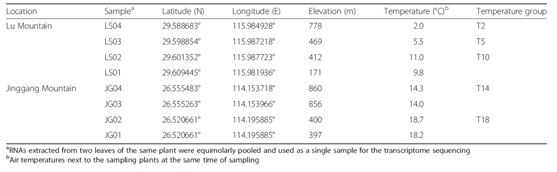
采样地点及时间:对庐山(2013年11月29日)和井冈山(2013年12月6日)共8处不同海拔位置的野生油茶叶片进行取样,并记录相应的经纬度、海拔及大气温度。
表1、野生油茶采样地点及分组情况
采样方法:选取开花期野生油茶植株进行取样,将3到5片完整叶片摘下用铝箔包好后,迅速放入液氮中,后转移至-80度冰箱保存。
RNA提取及转录组测序
油茶叶片用液氮研磨成粉,后取约100 mg粉末提取RNA。总RNA采用EASYspin Plus Plant RNA Kit (Aidlab,Beijing, China)提取。同一植株的两片叶片等量混合,最终根据取样时大气温度,8个样品分为5组T2,T5,T10,T14和T18用于测序(表1)。RNA-seq采用IlluminaHiSeq 2000双端测序(PE 2 x 100 bp)进行。
序列组装和Unigene注释
利用Trinity软件对测序获得的clean reads进行组装,组装最长的基因转录本当作unigene,将所有组装的unigene当作参考序列用于后续油茶叶片转录组分析。Unigene功能注释利用的数据库包括:Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG和GO等。
SSRs、SNPs和InDels检测
用MISA 1.0进行unigenes的SSRs检测,用Primer3进行SSRs引物的设计。用bowtie 2 (mismatch 0)对Clean reads进行参考unigene序列的比对,对比对的序列进行SAMtools和Picard的分类和重复去除。SNP和InDel利用GATK2进行识别,将QUAL<30.0和QD<5.0的进行去除。
遗传结构分析
为了说明不同纬度和海拔野生油茶样本的遗传结构,本研究筛选不超过2个等位基因位置和reads数不小于6个的SNP数据进行了系统进化分析。对不用样本进行SNP基因分型,利用MrBayes 3.2.5和FigTree软件构建系统进化树。
基因表达分析
利用RSEM计算每个样本中unigene的reads数并转换成FPKM,它的值作为不同样本unigene的基因表达水平。为了检验测序深度是否足够进行基因表达分析,不同比例比对上的reads被随机抽取进行评估,绘制的曲线说明了最终表达值10%以内的基因片段和比对上的read比例关系。如果曲线趋于平缓,则表明测序深度足够用来进行基因表达分析。
为了研究大气温度对基因表达模式的影响,用DESeq对不同温度分组间的差异基因表达水平进行FPKM密度分布比较分析,用分层聚类分析和韦恩图进行展示。根据unigene的功能注释,将预测有功能的差异表达基因(DEGs)用于冷适应性的候选基因的发掘。此外,通过比较DEGs与所有检测到基因表达的GO注释情况,弄清了涉及到冷适应的相关功能分组基因。
qRT-PCR分析
用qRT-PCR来检验转录组测序获得的DEGs的准确性。
研究结果:
测序及组装情况
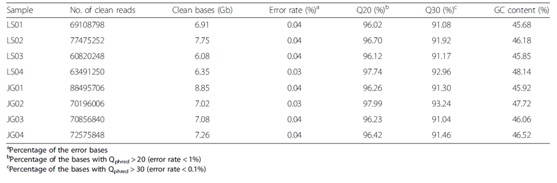
本实验共获得57.3 Gb高质量测序数据(表2),平均每个样本测序数据在6.08到8.85 Gb范围。测序数据组装了286121个转录本,长度从201到20507个碱基不等。最终获得的unigene的总长度为91.6 Mb。
表2、不同野生油茶样本测序数据统计表
Unigene功能注释
本研究对83352个unigene进行了成功注释(表3)。其中GO分析结果表明,生物过程相关基因最多,其次是细胞组分类。
表3、不同数据库对unigene的注释情况
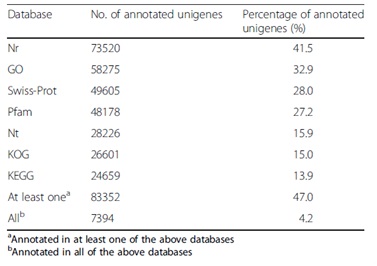
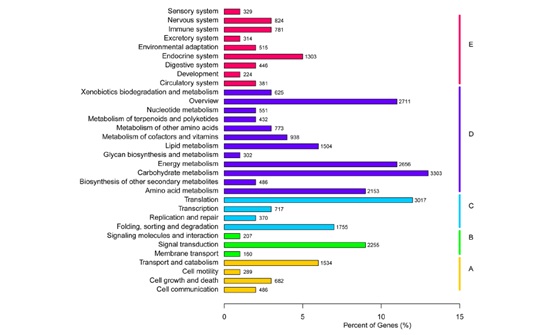
图1、KEGG通路富集分析
KEGG通路分类结果表明,注释结果涉及了不同代谢通路,其中碳水化合物代谢最多(图1)。
SSRs、SNPs和InDels检测
本研究共检测出25751个SSRs,SSR motif分布情况如图2所示,大约46.8%的SSRs是单核苷酸重复,主要是(A/T)n重复。第二多的是二核苷酸重复(37.2%),主要以(AG/GA/CT/TC)n为主,总共设计13962个SSRs的引物。针对分子标记开发目的,将一些复杂的SSRs,如(TA)6(TAC)6,(CT)8tatct(TC)6等和一些小于2个核苷酸重复的SSR去除,成功设计了7005个SSR引物。
本研究获得661280个SNPs,其中约54.%为非编码的SNPs。在编码区的SNPs里面,非同义与同义SNPs的比值为0.604。有103442个SNP位置在2个等位基因并且不小于6个比对上的reads中出现,这些为油茶的SNP分子标记开发提供有用信息。研究人员同时发现了47056个InDels,其中6534个可以用于后期分子标记开发。
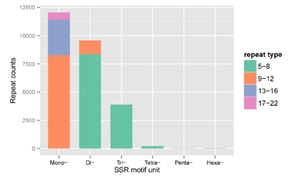
图2、SSR motifs分布图
遗传结构分析
研究人员随机选取9000个SNP用于系统进化分析。系统进化树构建结果如图3所示,除了JG01和LS02外,从庐山和井冈山取样的油茶是分开的,并且从高海拔取样在遗传上比两山之间差别更大。
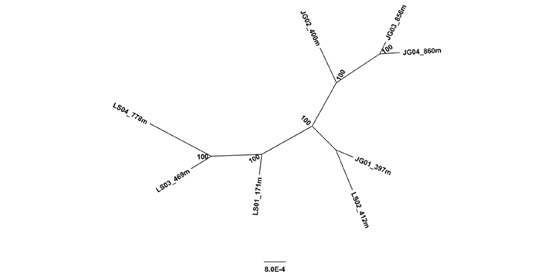
图3、不同地点取样油茶的系统进化分析
基因表达差异分析
根据FPKM密度分布图可以看出,当FPKM>3时,所有样本的曲线趋于饱和状态,这说明测序深度能够用于进行基因表达分析。不同温度分组的基因表达密度分布图如图4所示,基因表达模式可以根据相似性分为两组:T18、T14、T10代表的相对高温度组(10-18度)和T5、T2代表的相对低温度组(2-5度)。
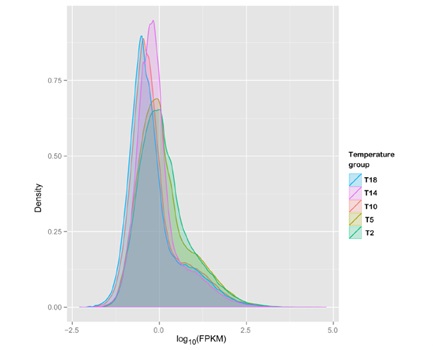
图4、FPKM密度分布图
差异表达基因的聚类热图表明,T18和T14聚在一起具有相对较高的表达模式相似性(图5)。同样的,基因表达模式随着大气温度的降低而变化,尤其是在T5和T2中上调表达的基因。
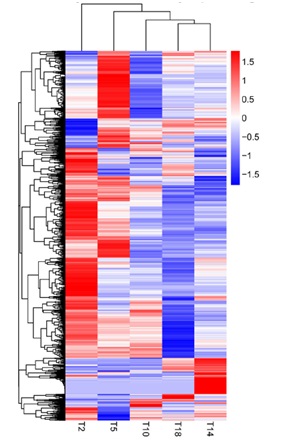
图5、差异表达基因聚类分析图
韦恩图对不同温度的差异表达基因数目差别进行了统计(图6)。
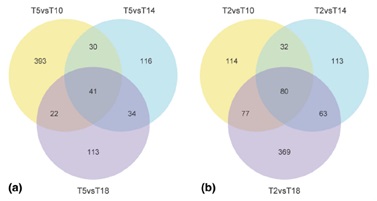
图6、差异表达基因韦尔图
经过进一步不同分组间的分析,鉴定了多组别间冷适应相关的候选基因。通过对这些差异表达基因分析,发现了多个SNPs,进一步分析非同义与同义SNP比值,推测部分差异表达基因可能处于正选择中。
对差异表达基因的GO分析表明,生物过程相关基因比较多。其中T2中的C重复结合因子(CBF)显著性表达上调,可能与冷适应相关(图7)。
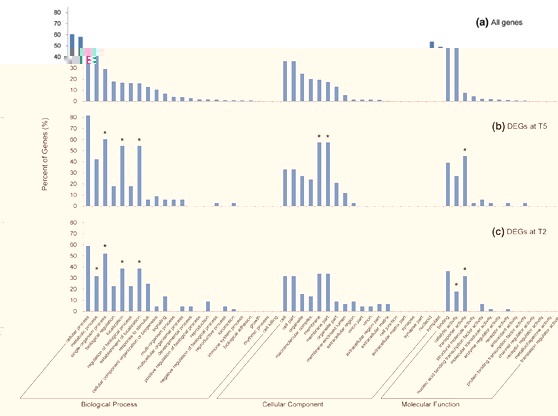
图7、差异表达基因的GO分析
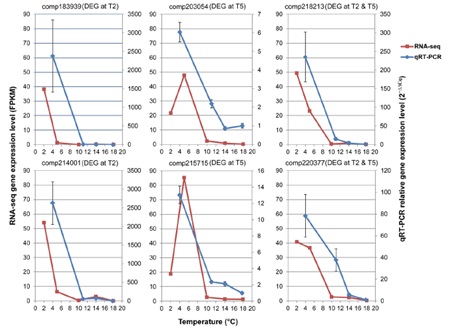
图8、差异表达基因在RNA-seq和qRT-PCR中的比对图
qRT-PCR验证分析
本研究对T5和T2中的糖转运基因差异表达进行了qRT-PCR验证分析,结果能够与RNA-seq结果很好的吻合,表明RNA-seq得到的差异表达基因结果是可信的。
总结:
l 实验共获得57.3 Gb高质量测序数据,组装了286121个转录本,获得的unigene的总长度为91.6 Mb。
l unigene GO分析结果表明,生物过程相关基因最多。KEGG通路分类结果主要涉及碳水化合物代谢等。
l 检测出25751个SSRs,以单核苷酸重复和二核苷酸重复为主。针对分子标记开发了7005个SSR引物。获得661280个SNPs,其中6534个可以用于后期分子标记开发。
l 相对较高温度组和相对较低温度组具有各自的基因表达模式。
关于天昊:
天昊生物具有对多倍体植物各种通量分子标记,如SSR和SNP精准分型的技术检测平台,2016年公司客户共发表SCI文章127篇,总影响因子达到404分。我们致力于为遗传学领域的研究者提供高质量的科研策略咨询、实验技术服务和遗传数据分析服务,期待成为大家科研工作中的“昊”助手与“昊”伙伴。