一、软件简介:
NTSYSpc是一款用于研究多元数据模式和结构的分子遗传研究常用分析软件。NTSYSpc原始版本于上世纪60年代开发出来,经过多年的更新现在NTSYSpc的功能已经非常强大,例如使用者可以通过邻接法或UPGMA方法构建聚类图来评估进化树情况,用于发现数据点样本的关联关系等,因此也常常用来进行SSR等分子标记的聚类分析。

软件网址: http://www.exetersoftware.com/cat/ntsyspc/ntsyspc.html
二、软件介绍:
软件界面
1、数据格式及文件输入:
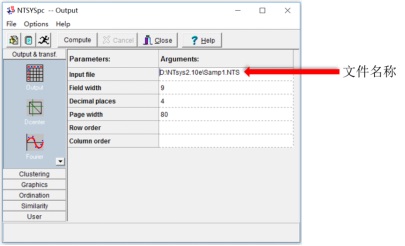
数据文件输入
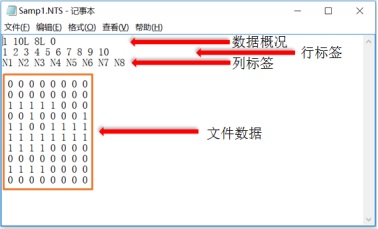
第一行为数据概况从左到右分别表示:1表示数据格式为rectangular data matrix;10L表示共10行(L表示行标签),8L表示共8列(L表示列标签),0表示无缺失。第二、三行数据代表具体的标签内容。文件主体为0、1数据矩阵。
2、相似性矩阵或遗传距离矩阵计算
在正确输入文件后,就可以对矩阵的相似性或遗传距离进行计算了,这也是进行后期聚类分析的基础。
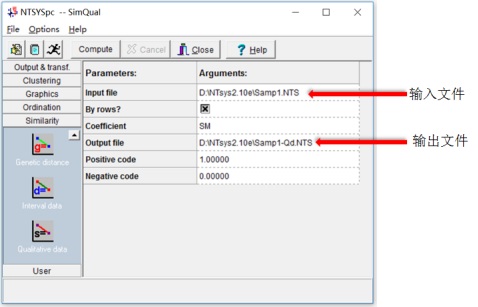
通常选择Similarity模块中的Qualitative data,在Input file中输入之前数据文件,如Samp1.NTS。如果个体是按行排列的,就要在By rows?进行勾选。如果个体是按列进行排列的,则不勾选。计算方法中矩阵系数coefficient有多种选择,如DICE,J,SM,PHI等,默认为SM。Output file输出文件可命名为Samp1-Qd.NTS,之后点Compute,得到相似性矩阵。对于其它一些定量数据数据则可采用simgend进行遗传距离计算,得到遗传距离矩阵。
3、聚类分析
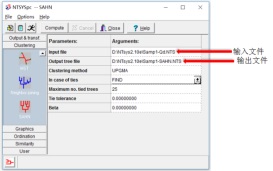
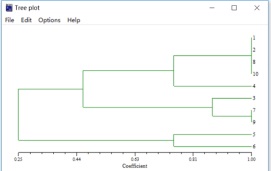
在得到相似性矩阵或距离矩阵文件之后,采用Clustering模块中的SAHN,Input file选择相似性矩阵文件Samp1-Qd.NTS,Output file输出文件命名为Samp1-SAHN.NTS,聚类方法中选择UPGMA,in case of ties选择FIND或者WARN,点击Compute就可以得到聚类结果。另一种常用的NJ聚类分析需要距离矩阵数据。在聚类分析之后,为了检验聚类结果的好坏,还可以进行相关性检验分析,这里就不再详述了。
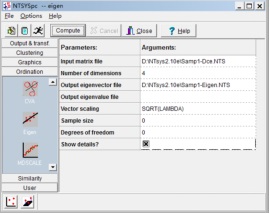
4、主成分分析
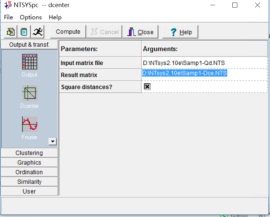
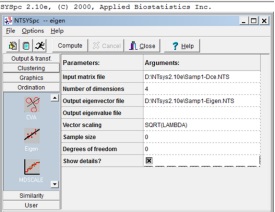
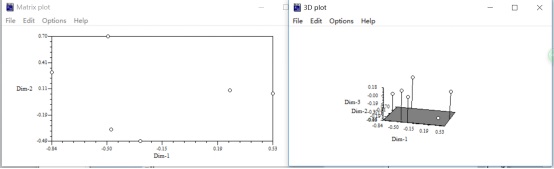
在进行主成分分析之前,需要首先对相似性或距离矩阵进行数据中心化处理,即在Output&transf模块中选择Dcenter命令,Input和Output中分别输入要分析的数据和结果文件的名称,点击Compute进行数据Dcenter转换。之后在Ordination模块中选择Eigen,选择要分析的数据中心化文件,点击Compute得到分析结果。分析完成后界面左下角会出现相应图标,点击即可查看二维和三维图形。
关于天昊:
天昊生物拥有多种SSR检测平台及SSRseqTM等专利技术,可以根据客户项目需求,提供不同数量样本和位点的高性价比微卫星检测服务。