英文题目: Multi-cohort analysis of colorectal cancer metagenome identified altered bacteria across populations and universal bacterial markers
期刊名:Microbiome 发表时间: 2018.4 影响因子: 8.496
背景:不同人群中肠道菌群的改变与结直肠癌(CRC)相关,目前发现几种细菌有助于肿瘤的发生,对于肠道微生物作为早期诊断标志物的潜在应用也有报道。然而,队列特异性噪声可能会扭曲CRC中微生物失调的结构,并导致研究结果不一致。本研究旨在探索不同人群间种水平上普遍改变的肠道微生物群落。
研究结果:
队列间微生物群落组成
我们积累了来自四个队列,美国(USA)、奥地利(AT)、中国(HK)、德国和法国(FD),包括271个对照组和255例CRC(人口统计学、临床和技术细节,如表1所示)的宏基因组测序序列。
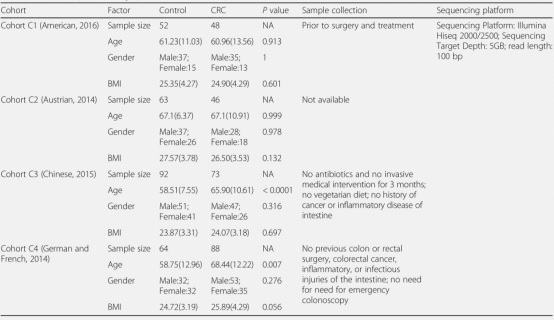
表1 粪便样品的人口学、临床和技术细节
USA、AT和FD队列中CRC病例和对照组间香农多样性指数差异不显著,然而,与HK队列对照组相比,病例组多样性指数显著降低。主坐标分析确定了不同群体间细菌组成存在显著差异(图1a: control; 图1b: CRC),还发现CRC病例和对照组间细菌总体组成有显著差异(图1c)。
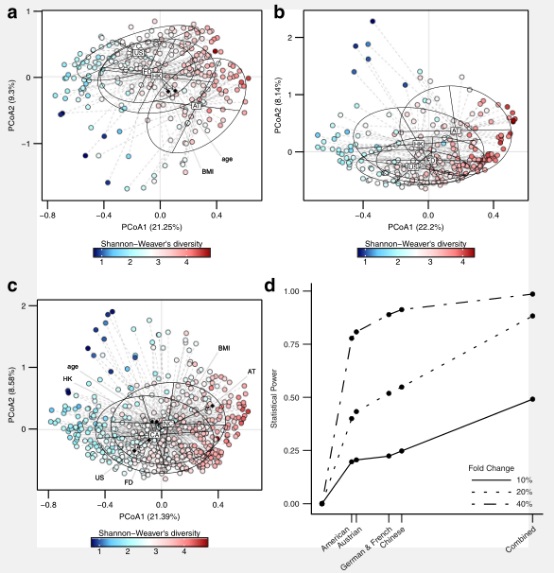
图1. 不同人群间微生物组成和统计差异。a-c:对照样本、CRC样本和所有样本各自的主坐标分析。d: 检测不同丰度细菌(不同细菌丰度变化程度(fold change=10, 20, 40%)vs队列样本大小(对照样本数×病例样本数))的统计功效。
队列间CRC和对照组丰度差异细菌
进行了模拟分析,比较了meta分析和单队列研究之间的统计功效。模拟分析表明,细菌丰度变化20%时估计功效为0.88;单一队列研究细菌丰度变化倍数为20%时估计功效则为0.5,这显示队列间优于单一队列研究(图1d)。利用meta分析来鉴定4个队列间CRC和对照组丰度差异细菌。使用秩和法,确定了7个CRC富集菌种和62个CRC减少菌种(图2a)。在这七个CRC富集菌种中, Bacteroides fragilis在所有四个人群中始终富集,而五个细菌(P. asaccharolytica, P. micra, P. intermedia, A. finegoldii, and T. acidaminovorans)在四个队列中的两个其丰富度表现出明显的变化(图2b)。在62个CRC减少细菌中有五种细菌被报道对健康有益,包括Clostridium butyricum, Streptococcus salivarius, Streptococcus thermophilus, Carnobacterium maltaromaticum, Lactobacillus gallinarum。为了确定这69个CRC相关细菌是否与CRC进程相关,我们研究了它们在CRC早期和晚期阶段中的丰度差异,3种细菌(Streptococcus sp. I-G2, Shewanella woodyi, Mycoplasma penetrans)对照组、早期CRC和晚期CRC呈下降趋势。
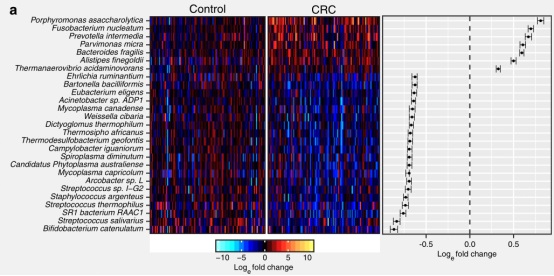
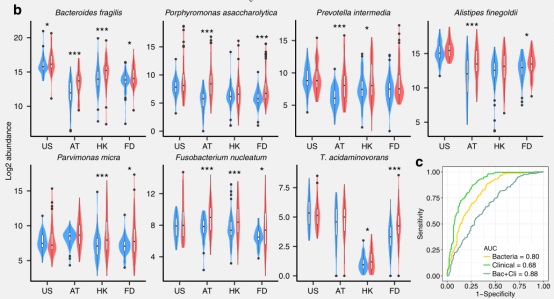
图2. 不同群体中CRC丰度差异的细菌。a 最大变化程度的7个CRC富集菌种和20个CRC减少菌种(左图), 个体配对变化程度的置信区间(右图)。b 7个CRC富集细菌在不同群体中的丰度。c 基于7个CRC富集菌种的SVM模型预测能力。
使用细菌标记物区分CRC和对照组
为了区分CRC患者和对照组,将七个CRC富含细菌物种装配到(SVM)模型中。获得的USA, AT, HK, FD 队列AUC曲线面积分别为0.83, 0.87, 0.84, 0.82。所有人群样本的AUC为0.80。当把临床表型信息(年龄,性别和体重指数)纳入以后,总的AUC增加到0.88(图2c)。七个CRC富集菌种在AT,HK和FD队列中区分早期CRC病例和对照的AUCs分别为0.84、0.82、0.84,提示其在早期CRC病例和对照之间的杰出分类性能。
CRC相关细菌之间的相关性
为了从生态学的角度深入了解细菌与细菌的相互作用,进一步研究。基于SparCC算法调查了CRC富集菌与CRC减少细菌之间的相关性。观察到CRC富集和减少的细菌分别形成了各自的共生网络,彼此呈负相关。有趣的是:CRC减少细菌中显著相关对的数量和相关强度在对照组高于CRC病例组(图3b)。CRC富集细菌和CRC减少细菌之间大多数是负的。七个CRC富集菌在CRC早期比CRC晚期的相关性更为密切,它们之间相关网络在CRC后期被破坏了。以加权程度为中心,发现梭状芽孢杆菌(Clostridium)在网络中中心性最高,这些中心物种可能在网络中起关键作用,与去除随机节点相比,当去除梭菌物种的节点时,网络连通性急剧下降。
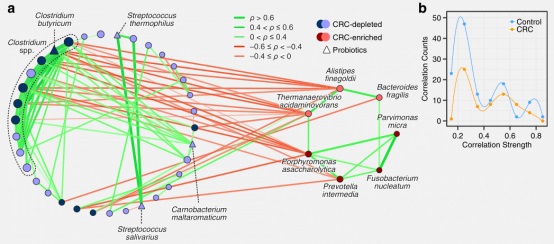
图3 CRC相关细菌之间相关性的Meta分析。a CRC样品中69个CRC富集或减少细菌之间的相关性。圆之间的相关节点用深蓝色标记。四个CRC富集的口腔菌种用暗红色标记,五个共生细菌以三角形表示,节点的大小与它们对应的中心性成正比。b CRC减少细菌的相关网络在对照组和CRC组之间的比较。
CRC富集和减少细菌相关的功能基因家族
将宏基因组序列映射到UniRef数据库,使用HUMAnN2将其分为10675个GO和8695 个 KO。共有311个GO分类和217个KO发现在CRC中富集, 31个GO和74个KO在CRC中减少。使用Spearman调查了七个CRC富集物种和GO/KO之间的相关性,确定了167个GO和143个KO与CRC富集细菌有显著的正相关(图4),我们定义了这些GO/KO为CRC富集细菌相关的GO / KO类别。为了研究相关的功能途径,我们将KO分类映射到KEGG通路,参与同一途径的KO被视为相关KO类别。鉴别了与癌症发展相关被多个KO共享的通路,发现了7个KEGG通路在CRC中富集(图4)。一些共生细菌被发现与CRC减少的GO/KO类别相关:Clostridium butyricum,Carnobacterium maltaromaticum与GO051606(刺激检测)密切相关;Streptococcus salivarius, S.thermophiles分别与K07104(邻苯二酚2,3-双加氧酶)和K07570(一般应激蛋白13)相关。
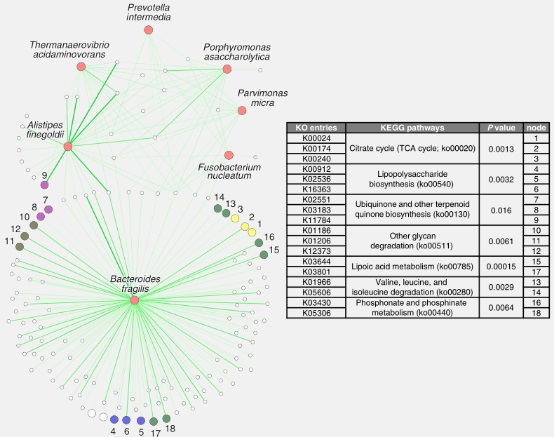
图4 CRC富集细菌与KO之间的相关网络。相同颜色的节点共享相同的CRC富集路径。
结果总结:
结论: