微卫星或简单重复序列(SSR)是基因组中信息非常丰富、用途也很广泛的一种遗传标记。然而传统的SSR分子标记开发常常是一项耗时费力及花费很大的工作。随着下一代高通量测序数据的快速积累,利用测序数据进行SSR分子标记挖掘便成了更加高效的方法。今年2月13日发表在Molecules上的文章“Mining and Development of Novel SSR Markers Using Next Generation Sequencing (NGS) Data in Plants”就这一问题进行了系统的梳理。

一、SSR的重要性及其作为遗传标记的应用
SSR是一个串联重复序列的亚类,由在所有原核生物和真核生物的基因组中发现的长度为1-6个核苷酸(基序)组成。在单个基因型中,由于SSR基序的串联阵列改变,重复单元的数量可能不同。因此,随着重复单元的增加,基因型的多样性也相应增加。同样,基序长度也影响重复的数量。
有大量SSR基因座分布在整个基因组中,特别是在真核生物的常染色质中,以及在编码和非编码细胞核和细胞器DNA中。由于微卫星信息丰富,突变率高,特异性强,种内多态性高,重复性好,易于数据化,多等位,跨分类群频繁出现特点,此外,SSRs的共显性特性允许直接测量杂合性,并且只需要少量DNA用于数据收集,因此微卫星得到了广泛应用。值得注意的是,它们被广泛应用于不同的目的,例如(1)遗传多样性;(2)发现数量性状基因座(QTL);(3)基因与标记连锁图谱的构建;(4)标记辅助选择所需性状(MAS);(5)法医学和亲子鉴定;(6)品种DNA指纹图谱;(7)全基因组关联研究;(8)基因流估计;(9)标记辅助育种(MAS);(10)单倍型测定;(11)杂种优势利用;(12)种质鉴定;(13)遗传诊断、转化体鉴定和细胞及组织鉴定。
SSR根据来源进行分类,主要分为基因组SSRs (g-SSRs)和表达序列标签SSRs (EST-SSRs),(注:还有一些其他命名方法,比如来源于核DNA的ncSSR,叶绿体DNA的cpSSR和线粒体DNA的mtSSR等)。EST-SSRs具有开发成本低、遗传多样性水平高以及向相关类群的转移能力强。相比之下,基因组SSRs由于引物结合位点的重复区域或简并性而具有较小的种间可转移性。尽管EST-SSRs的一个主要不足是在同一位点产生多组标记的序列冗余,但是这个问题可以通过将EST组装成单基因来解决。因此,EST-SSRs标记已经开发并在许多植物物种中使用,例如水稻、小麦、大麦、高粱、番茄、咖啡、橡胶、蓖麻和芝麻等。
二、SSR开发方法
SSR的开发可以依赖基因组DNA序列,也可以依赖由单链RNA (cDNA)合成的双链DNA,这取决于项目目标、未来的研究方案以及研究人员管理输出数据的能力。使用DNA直接测序更为直接,转录组测序(RNA-Seq )作为成功和有效的方法也可用于SSR挖掘,特别是用于没有参考基因组(从头组装)的植物(表1)。
表1、利用下一代测序技术开发的一些植物简单序列重复(SSR)标记列表
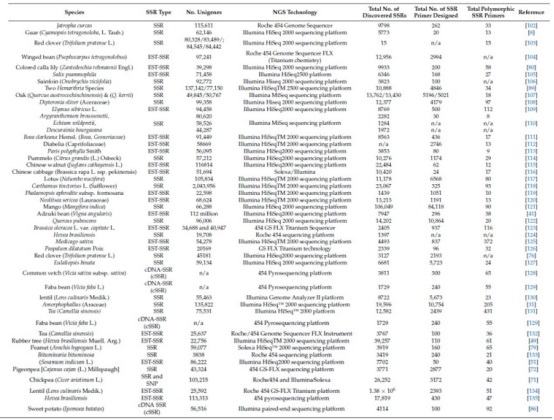
三、利用Illumina平台进行的转录组开发SSR过程概述
转录组从头组装过程包括RNA提取、cDNA文库构建、测序、数据过滤和质量控制、从头组装、单基因注释、SSR搜索和引物设计以及标记验证(图1)。
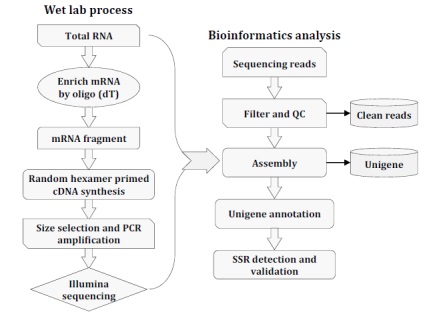
图1、从头转录组测序和组装过程的示意图
1、从头组装
用于从头组装RNA-Seq reads的工具有多种,例如Multiple-k、Rnnotator、Trans-ABySS、Velvet-Oases和SOAPdenovo-Trans。Trinity是一种近来越来越流行的转录组从头组装工具,它为序列读取生成单独的de Bruijn图。因此,每一个de Bruijn图指示了某一基因或基因座的转录复杂性,该基因或基因座被单独处理以获得全长剪接亚型,并梳理从同源基因提取的转录物。另外,Trinity先后应用了三个软件应用程序,即Inchworm,Chrysalis和Butterfly来管理大量的reads。该过程简要描述如下:
Inchworm:通过用最多的k-mers扩展序列,将reads组合成独特的转录本序列,然后只汇集不同剪接的转录本的特有部分。
Chrysalis:将Inchworm contigs按k-1重叠组成簇,为每个簇构建de Bruijn图组件,代表具有共同序列的一个或多个给定基因的完整转录情况。接下来,在簇之间划分完整的read集合。
Butterfly:并行独立解析拼接转录本,最终形成全长转录本。
Trinity产生的转录本应用于TGICL ( TIGR基因指数聚类工具)管线聚类的基因家族。此外,为了获得最终的单基因(如果有多个样品),TGICL将对每个样品的单基因再次计算,以获得最终的单基因(用于下游分析)。单基因将被分成包含多个相似度超过70 %的簇和单基因singletons(图2)。
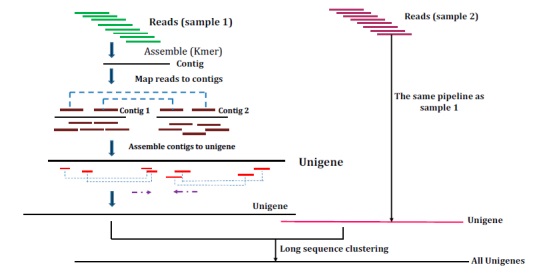
图2、转录组从头组装过程示意图
2、单基因功能注释
使用的功能数据库包括NCBI的非冗余核苷酸序列数据库(NT)和非冗余蛋白质序列数据库(NR)。此外,还包括Swiss-Prot、Pfam、KOG、GO和KEGG等数据库。所有数据库都使用Blast对齐组装的单基因,以获得每个单基因的注释功能。对于NR注释,可以使用Blast2GO或AmiGO获得单基因的基因本体注释。基因本体(GO)是一项重要的生物信息学联合项目,旨在解决在分子、细胞和组织系统级别上跨数据库生物功能的描述。
3、SSR挖掘和鉴定工具
为了在单基因中进行SSR挖掘和鉴定,人们开发出多种生信工具,比如MISA (MIcroSAtellite: http://pgrc.ipk-gatersleben.de/misa/)和SSRLocator (http://www.microsatellite.org/ssr.php)。然而,这些工具无法有效地处理大基因组序列,统计数据也不够准确。基于全基因组微卫星开发的分析工具—GMATo结果更快、更精确,可以针对任何大小基因组完成SSR分析。最近,研究人员开发了一款新的软件包GMATA,它通过映射和图形化的方式为快速SSR分析、标记开发和多态性筛选提供了新的策略和全面的解决方案,并将结果显示在具有其他基因特征的基因组浏览器中。此外,该软件还提供了高质量的统计图表。GMATA软件只使用侧翼序列作为设计PCR引物的模板,减少了计算内存,加快了大数据序列的设计过程。
4、DNA分离、PCR扩增和SSR验证
为了验证SSRs,需要提取DNA,合成目标SSR引物,通过PCR在不同植物品种或材料中进行扩增试验检测,最后选择成功的引物进行后续如遗传多样性研究等。
四、基于下一代数据中SSRs基因分型工具
最近,已经开发了许多软件工具来分析NGS数据中的SSRs,例如LobSTR、RepeatSeq、STRViper、STR-FM、PSR、rAmpSeq和STRScan。LobSTR运行时间快,在基因分型阶段考虑PCR stutter噪声。然而,对于单核苷酸SSRs和短于25bp的SSRs,LobSTR敏感性低。RepeatSeq工具是使用来自近交果蝇系误差分布图发布的。该工具利用其他程序绘制的读数,,并根据SSR基序、长度和碱基质量预测基因座最可能的基因型。然而,RepeatSeq的局限在于使用全部read作图法,这种方法在参考基因组中引入了对SSR长度的偏向,从而可能模糊真实的SSR变异谱。STR-FM (使用基于侧翼短串联重复的映射方法)被开发为用于从短读取测序数据中检测SSR并对其进行基因分型的灵活管线。另一种利用成对末端信息从深度测序数据中检测SSR变异的方法是STRViper。STRViper预测了基因组群体中的多态性重复序列,并发现了几个多态性重复序列,除了LobSTR使用自己的对齐工具之外,所有工具都需要预先对齐的数据。STRViper的性能在很大程度上取决于碎片大小的差异。
上述所有工具主要用于从SAM / BAM数据中分析SSR,它们从NGS数据中识别每个位点的gSSR等位基因。与上述工具不同,多态SSR检索工具(PSR)是为了从NGS数据中识别多态SSR而开发的,其中在非模式植物物种中,它们使用从头转录组作为SSR挖掘的第一序列资源,从而更有效地挖掘。2016年人们开发出了rAmpSeq重复扩增测序工具,适用于大多数物种的基因分型,使用低质量的DNA并产生多个标记,从而便于以每份样品更低的成本进行全基因组测序。另一个软件工具STRScan是为从基因组序列中生信挖掘SSRs而开发的,它比LobSTR和STR-FM具有更高的灵敏度。它在NGS数据中使用了一种特定的算法,对来自Sanger测序仪和Illumina测序仪的全基因组测序( WGS )数据进行有针对性的SSR分析。结果表明,STRScan可以在较短的计算时间内将目标集中被LobSTR遗漏的SSRs多达
关于天昊:
天昊生物具备完整的转录组(RNA-Seq)及全转录组检测服务产品线,同时拥有多种SSR检测平台及SSRseqTM等专利技术,可以针对客户具体项目需求,提供不同数量样本和SSR位点的高性价比SSR检测验证服务。