1.技术简介:
16S rRNA基因扩增子高通量测序,是通过对基因组目的区域进行PCR扩增,将目标区域DNA扩增富集后进行高通量测序的研究策略,可有效研究特定环境中的物种信息,检测几十至几百种微生物特性。近来,特定环境样本中某一类群微生物的绝对定量问题受到越来越多研究学者的关注,以往都是通过特定物种的qPCR绝对定量进行检测,但qPCR定量实验结果常常不稳定,且特定物种qPCR需要设计特定引物,对引物特异性要求较高,且引物优化难度较大,导致常规的qPCR绝对定量实验不再适用于组成较为复杂的环境样本微生物绝对定量。目前,16S rRNA基因测序手段通过某一OTU分类单元的序列数占总序列数的比值来获得相对丰度数据,然而相对丰度信息不能反映环境样本中物种绝对丰度情况,而基于16S spike-in standards的微生物绝对定量测序恰好能够获得环境样本中物种的绝对丰度数据。
该方法通过向样品DNA中添加一定量spike-in standards人工合 成序列,进行16S扩增子文库构建、测序,再根据spike-in standards 的16S扩增子reads数及其绝对拷贝数绘制标准曲线,计算出样品中OTU代表序列对应物种16S rRNA基因绝对拷贝数。添加16S spike-in standards到样品模板中进行16S rRNA基因扩增子高通量测序较好的解决了环境样本中微生物的绝对定量问题,同时也能得到环境样本中的物种组成信息。
说明:由于该方法需要额外添加外源核算序列,且对样本的DNA要求也较高,因此目前阶段只适合做一些常规的环境样本 (常规土壤样本,人粪便样本)。
2.技术优势
3.应用领域
4.技术参数
|
测序类型 |
推荐区域 |
样品要求 |
测序策略 |
数据要求 |
|
微生物16S扩增子绝对定量测序 |
V3 V4 V3+V4 V4+V5 |
样品类型:常规土壤样本和人粪便DNA,无明显降解; 样品需求量:总量>500ng;浓度≥20ng/μL; 纯度:OD260/280=1.7-2.0。 |
Illumina 2×250 Illumina 2×150 |
每个样本有效序列≥15万条,spike-in序列占总序列10%~50%;Raw data Q30数据达到70%以上,Clean data Q30数据达到80%以上。 |
5.技术路线
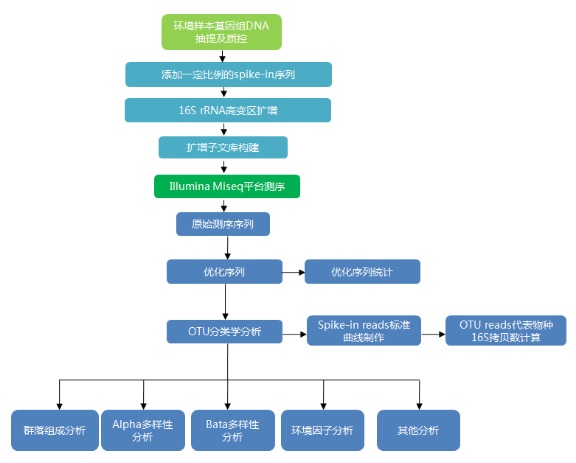
6.分析内容
|
标准分析 |
|||
|
数据统计与优化 |
OTU分类分析 |
||
|
原始序列数据统计 |
优化序列数据统计 |
OTU聚类分析 |
分类学分析 |
|
/ |
/ |
物种注释统计 |
进化树分析 |
|
|
|
OTU绝对定量 |
|
|
/ |
/ |
样本OTU Id对应的物种绝对拷贝数统计 |
|
|
群落组成分析 |
Alpha多样性分析 |
||
|
单样本物种组成饼图 |
单样本多级物种组成图 |
多样性指数分析 |
多样性指数差异分析 |
|
分类学系统组成树 |
物种总丰度柱状图 |
稀释性曲线 |
Shannon-Wiener曲线 |
|
丰度分布Bubble图 |
多样本群落结构柱状图 |
Rank-Abundance曲线 |
物种累积曲线 |
|
样本聚类树柱状图 |
Heatmap热图 |
/ |
/ |
|
Metastats分析 |
/ |
/ |
/ |
|
Beta多样性分析 |
/ |
||
|
Venn图分析 |
样本聚类树图 |
/ |
/ |
|
PCA分析 |
PCoA分析 |
/ |
/ |
|
NMDS分析 |
ADONIS分析 |
/ |
/ |
|
Lefse分析 |
/ |
/ |
/ |
|
高级分析项目 |
|||
|
环境因子分析 |
可选分析 |
||
|
Mantel test分析 |
Bioenv分析 |
PD分析 |
3D-PCoA分析 |
|
物种与环境因子相关性分析 |
RDA/CCA分析 |
PLS-DA分析 |
ANOSIM分析 |
|
/ |
/ |
Metagenomeseq分析 |
Wilcoxon秩和检验/ANOVA分析 |
|
/ |
/ |
PICRUSt功能分析 |
优势物种网络图分析 |
|
/ |
/ |
Indicator分析 |
OTU富集三元相图 |
7.服务流程
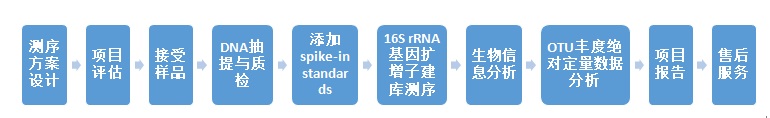
8. 结果展示
1) 样本OTU代表序列对应物种16S rRNA基因绝对拷贝数计算
a) 16S V4区扩增子测序的DNA模板中样品DNA和spike-in DNA添加情况
|
模板编号 |
模板中spike-in 拷贝数 |
模板中样本DNA量 |
样本信息 |
|
Y1_V4 |
2.67E+07 copies |
5 ng |
粪便样本 |
|
Y3_V4 |
2.67E+06copies |
5 ng |
土壤样本 |
|
Y4_V4 |
2.67E+07 copies |
5 ng |
淤泥样本A |
|
Y7_V4 |
2.67E+06copies |
5 ng |
土壤样本 |
|
Y42_V4 |
2.67E+06copies |
5 ng |
淤泥样本A |
注: Y4_V4和Y42_V4是同一个样本分别添加2.67E+07 copies和2.67E+06 copies的spike-in DNA后的混合样本。
b) 根据spike-in序列制作标准曲线(以Y4为例),计算优势OTU代表序列物种起始拷贝数:
表 1 Y4_V4 样品中spike-in序列数及拷贝数
|
spike-in序列编号 |
spike-in序列数 (OTUreads) |
spike-in 拷贝数 (copies) |
spike-in序列数取log log(OTUreads) |
spike-in 拷贝数取log log(copies) |
|
spike_1 |
68841 |
1.20E+07 |
4.837847 |
7.079061 |
|
spike_2 |
55078 |
1.20E+07 |
4.740978 |
7.079061 |
|
spike_3 |
9155 |
1.20E+06 |
3.961658 |
6.079061 |
|
spike_4 |
7949 |
1.20E+06 |
3.900312 |
6.079061 |
|
spike_5 |
1085 |
1.20E+05 |
3.03543 |
5.079061 |
|
spike_6 |
1034 |
1.20E+05 |
3.014521 |
5.079061 |
|
spike_7 |
72 |
1.20E+04 |
1.857332 |
4.079061 |
|
spike_8 |
66 |
1.20E+04 |
1.819544 |
4.079061 |
|
spike_9 |
66 |
1.20E+04 |
1.819544 |
4.079061 |
c) 以log(copies)为横坐标,log(reads)为横坐标,制作标准曲线如下:
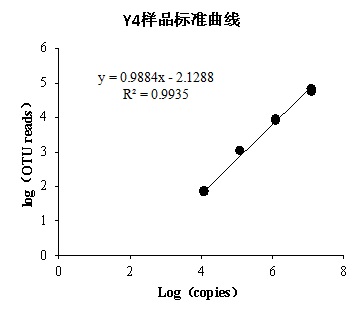
图 1 Y4样品的标准曲线
d) 标准曲线如下根据标准曲线计算的测试样品中优势OTU序列(Top10)的起始拷贝数:
表2 根据标准曲线计算的测试样品中优势OTU序列(Top10)的起始拷贝数
|
#OTU ID |
Y4_V4 OTUreads |
Y4_V4 copies |
Y4_V4 log(OTUreads) |
Y4_V4 log(copies) |
|
OTU60 |
47754 |
7.72E+06 |
4.67901 |
6.887707 |
|
OTU8 |
6052 |
9.55E+05 |
3.781899 |
5.980068 |
|
OTU73 |
3820 |
6.00E+05 |
3.582063 |
5.777887 |
|
OTU63 |
3650 |
5.73E+05 |
3.562293 |
5.757884 |
|
OTU72 |
3028 |
4.74E+05 |
3.481156 |
5.675795 |
|
OTU71 |
3017 |
4.72E+05 |
3.479575 |
|