Q1: 开发微生物16S扩增子绝对定量测序技术的初衷是什么?
A:
Q2: 目前qPCR技术在微生物定量研究中的短板是什么?
A: qPCR绝对定量技术是将已知拷贝数的标准品梯度进行稀释,进行qPCR检测,通过已知起始拷贝数的标准品可作出标准曲线,对样品进行目的基因qPCR检测,根据标准曲线就可以计算样品中物种的绝对拷贝数,虽然可以进行物种绝对定量分析,但是特定物种qPCR检测需要设计特定的引物,对引物特异性要求较高,且引物优化难度较大。
Q3: 目前常规16S扩增子测序技术在微生物定量研究中的短板是什么?
A: 常规16S扩增子测序技术是针对所有细菌共有的16S区域进行PCR扩增,将目标区域DNA扩增富集后进行高通量二代测序,然后将得到的序列与特定数据库比对,确认物种,通过OTUs聚类分析,就可以知道样品中的细菌结构组成和不同细菌的相对丰度, 虽然其具有高通量测序,大数据量、低花费,可客观还原菌群结构及相对丰度比例的巨大优势,但是其是通过某一OTU分类单元的序列数所占总序列数的比值来获得某个细菌的相对丰度比例信息,并不能反映样本中物种真实的绝对丰度情况,因此通过此技术得到的结果说X菌在A组相对于B组增多是错误的,应该是X菌在A组中的丰度比例相对于B组增多。
Q4: 微生物16S扩增子绝对定量测序相对于qPCR绝对定量技术和常规扩增子测序技术有什么明显优势?
A: 微生物16S扩增子绝对定量测序是一种将qPCR绝对定量技术和常规扩增子测序技术合二为一的一种创新性技术,可以说微生物16S扩增子绝对定量测序=常规16S扩增子测序+总细菌qPCR绝对定量+特定菌种qPCR绝对定量,因此通过微生物16S扩增子绝对定量测序既可以解析样本中总细菌的绝对拷贝数;解析样本中特定物种的绝对拷贝数;也可以进行Alpha多样性分析、群落组成分析、Beta多样性分析和环境因子分析等常规扩增子测序分析内容。
Q5: 微生物16S扩增子绝对定量测序的技术原理是什么?
A: 该方法通过向样品DNA中添加已知拷贝数spike-in人工合成序列,然后将样本进行16S扩增子文库构建、测序,再根据spike-in的16S扩增子reads数及其绝对拷贝数绘制标准曲线,根据OTU reads数,计算出样品中OTU代表序列对应物种16S rRNA基因绝对拷贝数。添加16S spike-in到样品模板中进行16S rRNA基因扩增子高通量测序较好的解决了环境样本中微生物的绝对定量问题,同时也能得到样本中的物种组成信息。
Q6: 目前微生物16S扩增子绝对定量测序技术的样本类型和样本量有哪些要求?
A:目前样品类型包括:常规土壤样本和粪便样本,其它样本类型则需要评估。如果客户直接送样本,要求:土壤≥5 g;粪便≥2 g;如果客户送样本DNA,要求:总量≥500 ng, 浓度≥20 ng/Μl, 纯度:OD260/280=1.7-2.0。
Q7: 直接送样本和送样本DNA有什么特别需要注意的事项?
A:因为客户提供的样本则包括样本或样本DNA,所以微生物16S扩增子绝对定量测序提供的菌种拷贝数展现形式统一是:16S基因copies/ ng DNA,而常规的菌种qPCR绝对定量文章在菌种拷贝数的展现形式上则是:16S基因copies/ g样本,因此为了将微生物16S扩增子绝对定量测序获得的16S基因copies/ ng DNA值能够对应到 16S基因copies/ g 样本上,需提前记录好以下信息:
Q8: 为什么微生物16S扩增子绝对定量测序技术每个样本有效序列≥15万条?
A: 这是因为spike-in序列会占用掉一部分测序数据量,为了保证样本常规的5万有效reads数据量要求,每个样本测序数据量需达15万有效reads。
Q9: 微生物16S扩增子绝对定量测序技术和qPCR绝对定量技术对比如何?
A:
准确度:我们比较qPCR绝对定量方法与16S扩增子绝对定量测序方法所测定的同一系列样本的细菌总拷贝数,Pearson相关性分析结果显示:两组数据在0.01水平上显著相关,相关系数为0.955,且两种方法计算的细菌总拷贝数值较为接近,未超过一个数量级,说明通过微生物16S扩增子绝对定量测序技术进行微生物绝对定量是可靠准确的。
稳定性:qPCR定量批次间及样品重复间(批次内)稳定性较差,而16S扩增子绝对定量测序目前的项目结果显示稳定性相对较好。
特异性:16S扩增子绝对定量测序所用的引物是常规16s 扩增子测序最公认的引物,引物特异性很好,并且这一对引物就可以得到总细菌和所有菌种的绝对拷贝数;而特定菌种qPCR检测则需要设计特定的引物,对引物特异性要求较高。
灵敏度:qPCR理论检测灵敏度可以达到101分子,实际检测中,有效灵敏度取决于标准曲线的菌种丰度下限,受qPCR检测平台和实验方法的限制,通常检测下限在102水平。16S扩增子绝对定量测序理论检测灵敏度为单分子,实际检测中,有效灵敏度取决于标准曲线的菌种丰度下限,与添加的spike-in总拷贝关联(添加105拷贝:检测下限为102拷贝,添加106拷贝:检测下限为103拷贝)。
Q10: spike-in序列的添加对样本原有群落组成和样本物种绝对丰度是否有影响?
A:
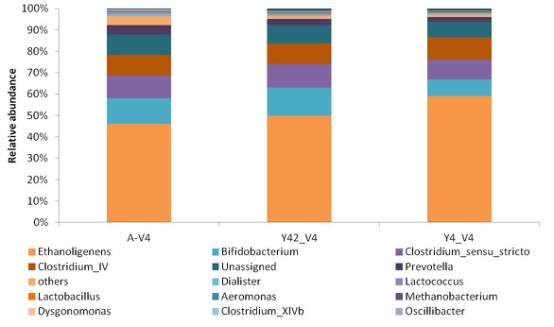
图1 属(genus)水平群落组成柱状图
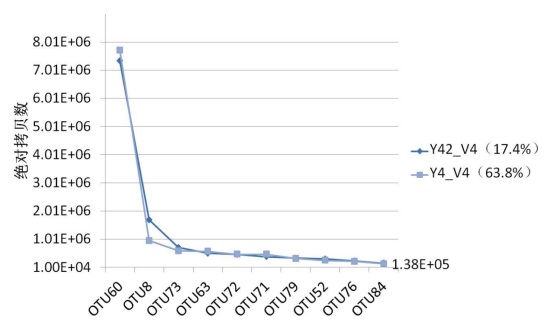
图2 优势OTU代表物种绝对拷贝数(Top 10 )
想了解更多“微生物16S扩增子绝对定量测序”内容,请点击链接: