近期Nucleic Acids Research发表了由实验结果支持的竞争性内源RNAs (competing endogenous RNAs,ceRNAs)数据库研究文章,重点介绍了LncACTda 2.0数据库的数据和功能更新,这为全转录组研究提供了强有力的工具支撑,下面我们就来看一下这个数据库概况。


网址:http://www.bio-bigdata.net/LncACTdb/index.html
摘要一览
LncACTdb 2.0是一个更新和显著扩展的数据库,提供了不同物种和疾病相关ceRNA的综合信息,成为研究ceRNAs的重要网络资源。具体包括:(1)从超过5000篇已发表文献中人工筛选到2663篇具有实验数据支持的ceRNA信息;(2)将数据库的范围扩大到23个物种和213种疾病/表型;(3)纳入更多的RNA类型,如环状RNA和假基因;(4)从TCGA数据中鉴定出33种癌症类型的候选lncRNA相关ceRNA的互作关系并对其评分;(5)为ceRNA提供存活率、互作网络和癌症标志的图解信息。此外,还开发了多种灵活的在线工具,包括LncACT-Get、LncACT-Function、LnCaCT-Survival、LncACT-Network和LncACTBrowser,用于进行定制分析、功能分析、存活分析、网络图解和基因组可视化。LncACTdb 2.0还提供了新设计的、用户友好的web界面来搜索、浏览和下载所有数据。BLAST界面便于用户通过输入自定义序列来查询数据集。热点界面为用户提供了他人研究最多的词条。
背景介绍
越来越多的证据表明,miRNAs受到存在有miRNA结合位点的长链非编码RNA(lncRNAs)、环状RNA(circRNAs)、假基因等的调控,从而竞争性影响了miRNA与天然靶标mRNA的结合,即竞争性内源性RNAs(ceRNAs)调控机制,它可以在不同的生理和病理过程中动态调节彼此间的表达。
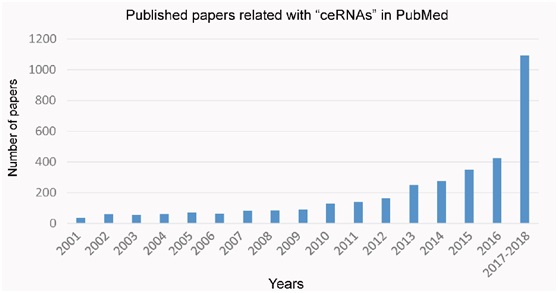
近年来发表的与ceRNA相关论文情况
到目前为止,人们已经建立了多个数据库,对miRNAs和其他分子之间的相互作用进行管理,如starBase v2、DIANA-LncBase v2、miRSponge和PceRBase等,它们为ceRNA的研究提供了重要资源。然而,这些数据库大都利用单一靶标方法预测相互作用,并且这些数据库中的物种只限于人类、老鼠和植物。除了miRSponge数据库存储了具有实验支持的11个物种的463个ceRNA关联外,尚无其他数据库专门用于收集、存储和分析具有实验结果支持的ceRNA信息以及综合注释。
改进的内容和新功能
为了满足这些需求,研究者在LncACTdb 1.0基础上发布了2.0版(LncACTdb 2.0),增加了更多数据和一些新功能(表1 )。
表1、LncACTdb 2.0内容统计列表
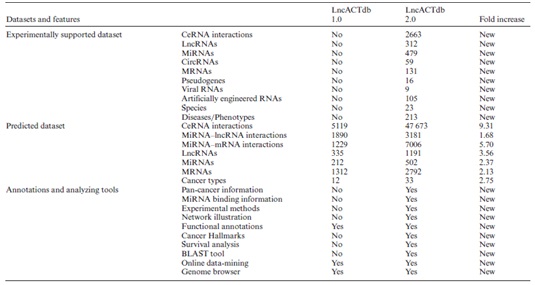
实验支持ceRNAs的新增条目
高置信度ceRNA信息是从文献中手动提取并集成到LncACTdb 2.0数据库中。在本次更新中,研究者通过使用与ceRNAs相关的关键词从PubMed检索了已发表的文献 (2018年10月之前)。按年份对5000多篇文章进行分类,经过人工判读汇总为有实验支持的数据集,如PCR、western印迹或荧光素酶报告分析数据,以及其他可靠的方法被考虑和进一步确定。目前,LncACTdb 2.0记录了总共2663个实验支持的ceRNA相互作用,包括312个lncRNAs、131个编码mRNAs、59个circRNAs和16个假基因。LncACTdb 2.0的范围扩大到23种和213种疾病/表型。
泛癌种lncRNA相关ceRNAs的扩展条目
研究者使用集成管线从TCGA中鉴定出候选的与lncRNA相关的ceRNA (图1)。使用四种具有严格阈值的miRNA靶预测方法(miRanda、RNAhybrid、TargetScan、PITA)来预测miRNA-lncRNA相互作用。此外,从starBase v2下载的41个AGO-CLIP-seq数据集被整合到管线中,以识别lncRNA序列上实验支持的miRNA结合位点。BEDTools使用重叠长度>1作为阈值,比较CLIP-seq峰对应基因组坐标和预测的miRNA结合位点。miRNA-mRNA调控关系数据来源于通过荧光素酶分析、PCR和Western blot等实验方法验证的TarBase (v8)和mirTarBase (v2018)。如果一个lncRNA和mRNA与同一个miRNA相互作用,这个lncRNA-miRNA-mRNA竞争三联体被称为候选ceRNA相互作用。功能性ceRNA则定义为:corr(lncRNA,miRNA )<0,corr(mRNA,miRNA ) <0和corr(lncRNA,mRNA )>0,其中corr(a,b )分别代表基于其表达值的基因a和b的皮尔逊相关系数。在LncACTdb 2.0中,癌症类型已经从12种扩大到33种。最终,LncACTdb 2.0中发现了47673个跨癌种功能性ceRNAs。为了促进ceRNAs的研究,LncACTdb 2.0提供了miRNA-lncRNA相互作用的miRNA结合位点的详细信息,以及miRNA-mRNA相互作用的实验验证方法。
网络、功能、标志和预后的扩展内容
对于每个与lncRNA相关的ceRNA条目,LncACTdb 2.0构建了一个由ceRNA及其相关的竞争邻组构成的网络,并进一步提供了图解。LncACTdb 2.0中使用了“按关联定罪”策略来执行ceRNAs的功能注释。对于路径的注释,从MSigDB下载了总共1329条路径,包括KEGG、BioCarta、Reactome、PID、STKE和SIG。收集Entrez IDs作为功能基因列表。对于GO注释,总共收集了5917个代表功能术语的基因集合。每个GO术语中的Entrez IDs被用作功能基因列表。已经确定促进肿瘤生长和转移的癌症标志过程的基因组从MSigDB下载。为了进行生存曲线分析,收集了来自TCGA的10141名患者的临床随访信息。根据ceRNAs表达值的线性组合,用Cox回归系数加权,构建了风险评分模型。此外,中位或平均风险分数被用作一个临界值,将患者分成两组,两组患者有不同的生存风险。对两组患者进行Kaplan-Meier生存分析,并使用对数秩检验评估统计学意义( P <0.05 )。
用于数据发现和分析的新开发工具
高通量技术产生了大量的表达谱信息,迫切需要通过分析这些数据集来解析疾病病理和发现癌症生物标志物。在LncACTdb 2.0中,研究者更新了LncACT-Get工具,让用户根据定制的输入识别新的ceRNA关系。用户可以上传某一疾病或表型的表达谱,LncACT-Get实现管线集成,以识别具有相应活动评分和P值的功能性ceRNA。为了研究受lncRNAs影响的下游生物过程,LncACT-Function工具被开发出来。它基于“按关联定罪”策略对用户输入的lncRNAs进行功能分析。LncACT-Function收集了成千上万条路径和生物术语作为功能背景。为了发现新的ceRNA预后生物标志物,研究者开发了LncACT-Survival工具,该工具对33种癌症类型的TCGA中的ceRNA相互作用进行在线生存分析。此外,LncACT-Survival工具还提供单个lncRNA、miRNA或mRNA的生存分析。为了便于ceRNA网络的可视化还开发出LncACT-Network工具。对于定制的lncRNA或mRNA,LncACT-Network工具将提供所有可能的ceRNA交互的全局视图,以及不同ceRNA之间的更多的cross-talk信息。
更灵活的访问数据集方式
LncACTdb 2.0为数据发现和访问提供了更灵活的方式:(1)开发了快速搜索引擎,允许用户搜索实验支持和预测的数据集。输入的关键词可以是任何一种基因克隆、miRNAs、mRNAs、circRNAs、假基因、疾病、细胞系、主要位点等。(2)开发了名为BLAST的新数据访问工具,以实施定制的排序搜索。用户可以输入RNA序列,以便识别相关的ceRNAs。(3)热点界面显示LncACTdb 2.0的访问记录,向用户提供其他研究人员研究最多的项目。(4) LncACTBrowser是一个基于网络的基因组浏览器,动态显示ceRNAs的不同记录。它提供了全面的信息跟踪,包括参考序列、转录本、miRNA结合位点(由miRanda、TargetScan、PITA和RNAhybrid方法预测)和CLIP-seq峰(41个数据集)。(5)通过所有查询步骤,点击“复制”、“Excel”和“CSV”按钮,可以灵活下载结果。
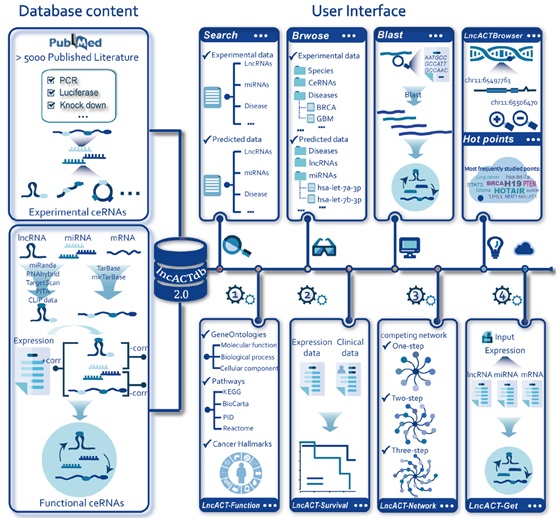
图1、LncACTdb 2.0数据库内容及用户界面。左侧是数据库内容,包括从低通量和高通量实验中鉴定的ceRNA信息。右侧是LncACTdb 2.0的用户界面。在此界面中,搜索、浏览、BLAST和LncACTBrowser模型提供了访问数据集的灵活方式。已经开发了在线工具包括LncACT-Function、LncACT-Survival、LncACT-Network和LncACT-Get,以执行定制分析和数据可视化。
数据库构建和改进的用户界面
LncACTdb 2.0中的所有数据都由MySQL数据库记录和管理。web服务器是通过使用Tomcat容器中的Java服务器页面更新的。LncACTdb 2.0提供了一个用户友好的web界面,用户可以通过几个简单的步骤搜索、浏览、分析和下载数据(图2)。作为在搜索界面中输入的lncRNA MALAT1的一个例子(图2A),所有可能的ceRNA都将显示在结果页面中(图2B)。为了过滤出有趣的ceRNA,用户可以通过点击不同列的标题来重新排序结果表格。第一栏将引导用户进入ceRNA的详细信息页面。LncACTdb 2.0提供了综合信息,包括基本信息、泛癌信息、MALAT1相关ceRNA的预测和实验信息(图2C)。为了进一步分析数据集,开发的几个在线工具,可以在每页的导航栏上轻松访问(图2E-H)。LncACT-Function工具根据GO术语、路径和癌症特征对MALAT1进行功能分析(图2E)。LncACT-Survival工具执行生存分析,并为ceRNA交互提供Kaplan-Meier生存曲线(图2F)。LncACT-Network工具提供了所有可能相关ceRNA交互的全局视图(图2G)。用户可以通过调整不同步骤来重置网络规模。根据定制的表达式配置文件,LncACT-Get工具实现了一个集成管道,以识别功能性ceRNA以及相应的活动分数和P值(图2H)。此外,LncACTdb 2.0提供了更灵活的方法来访问数据集。一个浏览页面被设计用于根据不同的分类对数据库进行全面的浏览(图2D)。热点页面提供了人体地图插图和其他研究人员研究最多的项目(图2I)。BLAST页面实现定制的排序搜索。用户可以输入新的RNA序列,以便识别相关的ceRNAs (图2J)。LncACTBrowser是一个基于网络的基因组浏览器,它提供全面的轨迹,包括参考序列、转录本、miRNA结合位点和CLIP序列峰值信息(图2K)。

图2、使用LncACTdb 2.0的案例研究和工作流程。(A)搜索模块与MALAT1示例的界面。(B) MALAT1的搜索结果,包括预测和实验支持的数据集。(C)带有详细信息的搜索结果页面。(D) LncACTdb 2.0的浏览界面。(E)基于GO术语、途径和癌症特征的MALAT1功能分析。(F)与MALAT1相关的ceRNAs的生存分析和Kaplan-Meier生存曲线。(G) MALAT1的所有可能相关的ceRNA交互的全球视图。(H) LncACT-Get工具实现了一个集成管道,以根据定制数据识别ceRNA的功能交互。(I)热点页面提供了人体地图和其他研究人员研究最多的项目。(J) BLAST接口实现定制的测序搜索,以识别相关的ceRNAs。(K) LncACTBrowser提供MALAT1的全面基因组信息,包括参考序列、转录本、miRNA结合位点和CLIP-seq峰。
研究人员预测,在未来的LncACTdb数据库版本中,通过高置信度实验或高通量分析确定的ceRNA数据集将继续快速增长。不断更新的LncACTdb数据库,增加数据集和功能界面,这将提高人们对复杂疾病中编码和非编码RNA的理解。
全转录组测序技术优势:
• rRNA去除建库,保留了完整的RNA种类信息;
• 链特异性文库,可以保留转录本的链信息,更准确地检测反义RNA;
• 使用高通量测序,能够获得更加全面的RNA信息,包括低丰度的RNA;
• 通过测定的序列信息精确地分析不同类型RNA的表达丰度变化及其生物学功能;
• 分析同一样本中的mRNA、lncRNA、miRNA和circRNA四种类型RNA,明确这些RNA之间的共表达和调控关系,揭示ceRNA调控机制。
关于天昊:
天昊生物具有丰富的转录组和全转录组测序经验,我们致力于为研究者提供高质量的科研策略咨询、实验技术服务和遗传数据分析服务,期待成为大家科研工作中的“昊”助手与“昊”伙伴。欢迎联系我们具体咨询!邮箱:techsupport@geneskies.com 电话:400-065-6886