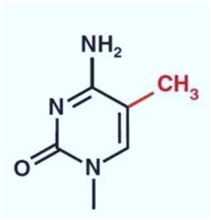
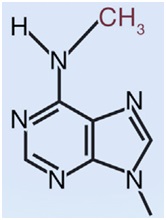
DNA甲基化是表观遗传学领域一个重要的研究方向,真核生物中最常见的DNA修饰非5-甲基胞嘧啶(5mC)莫属了,这也是我们研究最为广泛的一种修饰方式(图1)。然而在原核生物中最常见的DNA修饰方式则为N6-methyladenine (6mA),即腺嘌呤第6位氮原子甲基化修饰(图1)。那么在真核生物生物中是否也存在6mA的修饰呢?这个问题在早期一直存在争论。
随着6mA在原核生物中重要的功能(保护DNA、参与DNA复制和修复、基因调控等)被揭示,真核生物中6mA的研究也逐渐增多,早期主要集中在单细胞原生生物中,如四膜虫、草履虫和衣藻等,6mA大约占据这些基因组所有腺嘌呤的0.4-0.8%。而最新的报道显示6mA的修饰发生在更高等的生物中,如线虫、果蝇、蚊子和植物等。在人类基因组中是否也存在6mA的修饰呢 ADDIN EN.CITE ADDIN EN.CITE.DATA 1,2?本文针对human样本中发生6mA修饰的6篇文章进行了详细整理。
|
No1. DNA 6mA去甲基化酶ALKBH1增强了人间充质干细胞的成骨分化 3,Bone Research, 2016, IF: 12.354


研究发现了间充质干细胞ALKBH1的表达水平在成骨诱导过程中是上调的,而敲低ALKBH1增加了6mA的水平,同时显著降低了成骨相关基因的水平。体内实验也显示ALKBH1的缺失限制了骨头形成能力。而过表达ALKBH1能够增强成骨细胞的分化。机制上,ALKBH1缺失会导致ATF4启动子区域6mA修饰的积累,从而引起ATF4转录沉默;而ATP表达的重建能够成功挽救成骨分化。
从这篇文章中能够发现6mA修饰在干细胞分化表观调控中可能也发挥了重要作用,这样的调控机制和研究思路是不是可以借鉴?
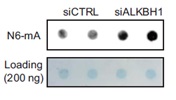
Dot Blot实验显示敲低ALKBH1显著增加了6mA的水平
No2. 利用单分子实时测序比对表征真核生物基因组6mA修饰 ADDIN EN.CITE ADDIN EN.CITE.DATA 4,Genome Research, 2018年5月,IF: 10.101

从文章标题能够看出本研究内容比较明确,利用三代单分子实时测序技术(SMRT)对两种真核生物基因组进行测序,在衣藻中构建了第一个完整的6mA单碱基和单分子分辨率的图谱;在人类淋巴母细胞中,联合SMRT和独立的测序数据推断6mA修饰富集在早期全长的LINE-1原件启动子区域。
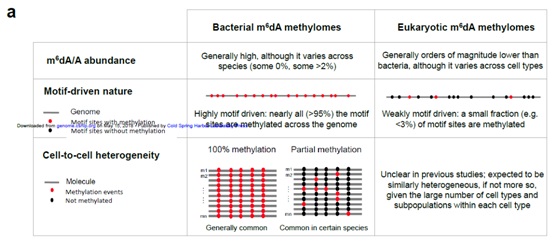
细菌和真核生物6mA甲基化组之间的差异
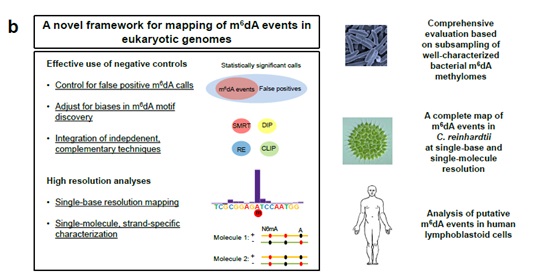
新的方法比对真核生物基因组中6mA修饰
No3. 人类基因组中6mA DNA修饰 ADDIN EN.CITE ADDIN EN.CITE.DATA 5,Molecular Cell, 2018年7月,IF: 14.248

看这个高大上的名字就知道这篇文章把矛头直指人类样本中6mA修饰,作者利用多种方法表征了人类基因组中6mA修饰,包括PacBio SMRT, 6mA-IP-seq, LC-MS/MS, 6mA-IP-qPCR等,可以说是第一篇paper系统地揭示了人类基因组中6mA修饰图谱。
其中发现6mA广泛存在于人类基因组中,一共有881,240个6mA位点,占据所有腺嘌呤约0.051%,[G/C]AGG[C/T]是6mA修饰最显著相关的motif。在人类细胞样本中6mA位点富集在编码区,而且标记了活跃转录的一些基因。
人类基因组中DNA 6mA和N6-去甲基化腺嘌呤分别被N6AMT1和去甲基化酶ALKBH1调控。而癌症中6mA丰度显著更低,伴随着N6AMT1水平下降,ALKBH1水平上调;6mA修饰水平的下调能够促进肿瘤形成。
因此这篇报道比较系统地阐述了人类基因组中的6mA修饰及6mA修饰在肿瘤形成中的重要作用!
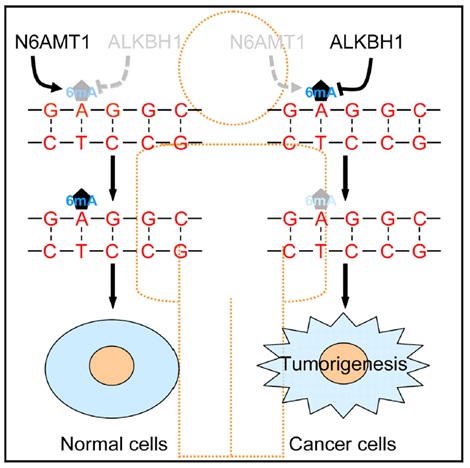
N6AMT1和ALKBH1在人类正常细胞和癌细胞中重要功能示意图
No4. 人线粒体基因组上6mA单碱基分辨率测序发现链非对称性cluster与SSBP1有关 ADDIN EN.CITE ADDIN EN.CITE.DATA 6,Nucleic Acids Research, 2018年10月,IF: 11.561

作为NAR突破性文章,作者首先建立了6mA-交联-外切酶-测序(6mACE-seq)的方法在单碱基分辨率下检测基因组范围内的6mA,在合成的DNA及细菌基因组中证实了方法的准确性。同时利用这项技术绘制了人类基因组6mA图谱,准确地重现了已知的6mA富集在活跃的逆转录转座子上,而线粒体6mA cluster不对称地富集在重链上。
研究人员同时发现了一个新的6mA结合蛋白SSBP1,一种已知的覆盖重链的线粒体DNA复制因子,将6mA和线粒体DNA复制调控联系在一起。最后也表征了ALKBH1作为一种线粒体蛋白与6mA去甲基化活性有关,ALKBH1的缺失降低了线粒体氧化磷酸化。
因此这篇报道主要揭示了6mA在人类线粒体中的重要功能!
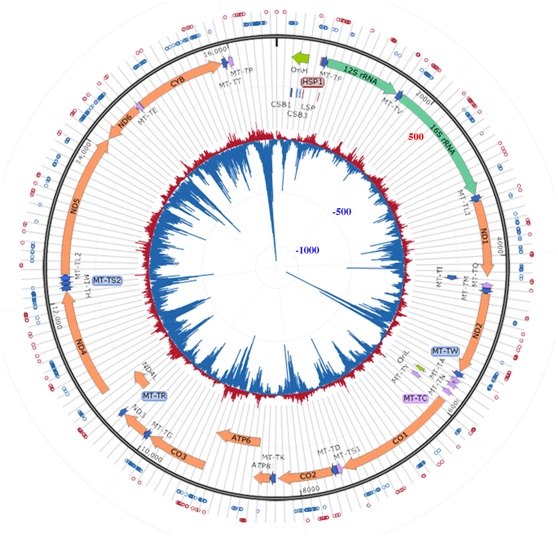
人类线粒体6mA图谱
No5. 恶性胶质瘤中6mA DNA修饰 ADDIN EN.CITE ADDIN EN.CITE.DATA 7,Cell, 2018年11月,IF: 31.398

标题言简意赅,在恶性脑部肿瘤--胶质瘤中表征6mA甲基化修饰,Cell主刊上发表的文章,内容定会详实:恶性胶质瘤中6mA的水平呈现明显的上调,而且与异染色质组的蛋白修饰共定位,特别是H3K9me3。6mA水平受到DNA去甲基化酶ALKBH1的调控,其缺失通过降低染色质可接近性导致oncogenic通路转录沉默。而对患者来源的胶质瘤模型靶向ALKBH1能够抑制肿瘤细胞的增殖,并且延长了移植瘤小鼠的生存时间,这表明这种新的DNA修饰可能是胶质瘤潜在的治疗靶点。
又一篇在癌症中验证了6mA修饰的重要功能!
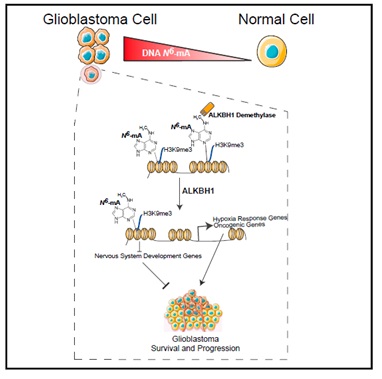
ALKBH1及6mA在恶性胶质瘤中重要功能示意图
No6. 利用合成的读-写(Read-Write)模块构建表观遗传学调控 8,Cell, 2019年1月,IF: 31.398

从标题上看文章挺有意思,研究团队在细胞样本中利用6mA修饰构建了一个正交的表观调控系统,利用合成的因子去write and read 6mA,因此招募转录调控因子控制报告位点。并且利用该系统和数学模型构建了调控回路,诱导基于6mA的转录状态,促进了其空间传播,维持了这些状态的表观遗传记忆(仅看这些文字确实懵逼,详见文章)。
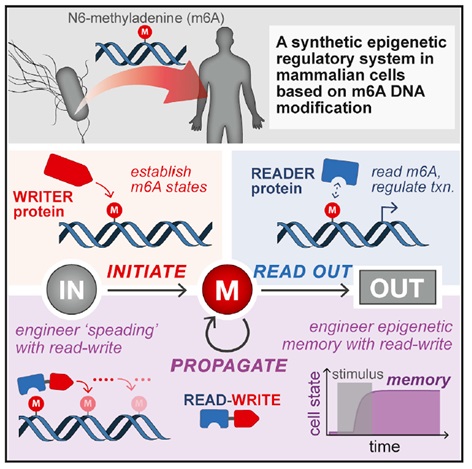
合成的6mA表观遗传调控系统
|
参考文献:
ADDIN EN.REFLIST
1. Heyn, H. & Esteller, M. An Adenine Code for DNA: A Second Life for N6-Methyladenine. Cell 161, 710-3 (2015).
2. Luo, G.Z., Blanco, M.A., Greer, E.L., He, C. & Shi, Y. DNA N(6)-methyladenine: a new epigenetic mark in eukaryotes? Nat Rev Mol Cell Biol 16, 70510 (2015).
3. Zhou, C., Liu, Y., Li, X., Zou, J. & Zou, S. DNA N(6)-methyladenine demethylase ALKBH1 enhances osteogenic differentiation of human MSCs. BoneRes 4, 16033 (2016).
4.Zhu, S. et al. Mapping and characterizing N6-methyladenine in eukaryotic genomes using single-molecule real-time sequencing. Genome Res 28, 1067-1078(2018).
5. Xiao, C.L. et al. N(6)-Methyladenine DNA Modification in the Human Genome. Mol Cell 71, 306-318 e7 (2018).
6.Koh, C.W.Q. et al. Single-nucleotide-resolution sequencing of human N6-methyldeoxyadenosine reveals strand-asymmetric clusters associated with
SSBP1 on the mitochondrial genome. Nucleic Acids Res 46, 11659-11670 (2018).
7.Xie, Q. et al. N(6)-methyladenine DNA Modification in Glioblastoma. Cell 175, 1228-1243 e20 (2018).
8. Park, M., Patel, N., Keung, A.J. & Khalil, A.S. Engineering Epigenetic Regulation Using Synthetic Read-Write Modules. Cell 176, 227-238 e20 (2019).