t-分布随机邻域嵌入(t-distributed stochastic neighbor embedding, t-SNE)广泛用于单细胞RNA测序(scRNA-seq)数据可视化分析,但是它很难扩展到大型数据集。2019年2月11日发表在Nature Methods的最新文章,推出了一种基于快速插值t-SNE的单细胞RNA-seq数据分析方法,该方法极大地加快了t-SNE的分析速度,消除了数据向下采样(downsampling)的要求,更易对稀有细胞群体进行可视化。此外,本研究基于一维t-SNE实现了scRNA-seq热图的可视化,以便同时对成千上万个基因的表达模式进行可视化。
软件下载网址:https://github.com/KlugerLab/FIt-SNE或
https://github.com/KlugerLab/t-SNE-Heatmaps

什么是t-SNE:即t-分布随机邻域嵌入算法,是目前一个非常流行的对高维度数据进行降维的算法, 具体说来就是把数据集中数据之间的高维欧式距离转变了条件概率来表示数据之间的相似度,由Laurens van der Maaten和 Geoffrey Hinton于2008年提出(原文链接:http://jmlr.csail.mit.edu/papers/volume9/vandermaaten08a/vandermaaten08a.pdf),它可以有效的将高维度数据转换成二维图像,目前已成为单细胞RNA测序(scRNA-seq)数据可视化最常用的一种分析内容。
scRNA-seq可以在单细胞水平进行高通量转录组分析,并越来越多地应用于研究生理和疾病过程中细胞间的异质性问题。scRNA-seq数据的可视化,对于分析已知细胞群体中不同标记基因的表达和识别新细胞类型方面发挥了关键作用。在过去十年中,使用t-SNE的数据可视化已经成为scRNA-seq分析的基石。t-SNE用于将scRNA-seq数据集嵌入到低维空间中,使得单细胞数据的高维转录组空间与低维空间中保持一致。嵌入通常由感兴趣基因的表达水平来决定并进行颜色标注,一次对一个基因进行分析。
目前t-SNE应用于scRNA-seq数据分析存在的问题
由于scRNA-seq实验技术的快速发展,需要分析的细胞数量呈指数增长。最近的研究检测数据集已经超过了100万个细胞中的30000个基因的表达。对如此大量的细胞进行分析有助于对较小样本中不明显的罕见和中等大小的亚群进行表征。然而,用于构建t-SNE嵌入的现有算法在计算上是成本很高的,通常需要在t-SNE之前对细胞进行向下采样来降低计算量,但这又会导致错过罕见的细胞群体。此外,去除可能表达给定标记基因的少数细胞可能会使中等大小的群体难以识别。
将t-SNE应用于scRNA-seq数据的另一个困难是,由于每个数据集有大量的标记基因,将标记基因的表达水平叠加在单独的二维(2D) t-SNE图上很麻烦。实际上,只有少量这样的图可以在视觉上进行比较。
本研究对t-SNE的改进
本文对t-SNE在scRNA-seq数据可视化中的应用提出了两点改进。首先,研究者提出了基于快速傅立叶变换(FFT)加速插值的t-SNE (FIt-SNE),这是一种基于多项式插值的一维(1D)和二维t-SNE快速计算算法,并使用FFT进一步加速。本研究还展示了t-SNE热图,这是一种基于1D t-SNE的热图可视化方法,它同时可视化成百上千个基因的表达模式。
研究者使用FIt-SNE嵌入了一个由130万个小鼠脑细胞组成的数据集,并从Allen Brain Atlas中成功识别出两种已知的细胞类型,而这两种细胞类型无法使用50000个细胞的随机子集来识别(图1),因为该子集没有足够的细胞同时表达Sncg和Slc18a8两种标记。

图1、通过标记基因识别大数据集中的亚群。a)使用FIt-SNE分析130万小鼠脑细胞的完整数据集。b)向下采样到随机的50000个细胞结果。
使用t-SNE探索scRNA-seq数据,每个图都被不同标记基因的表达模式所着色。人们通常最感兴趣的是哪些基因与哪些簇相关联,而不是簇的形状或相对位置。已有研究表明,t-SNE保留了良好聚类数据的聚类结构,而与嵌入维度无关,因此一维t-SNE(1D t-SNE)通常包含与二维t-SNE(2D t-SNE)相同的信息。因此本研究者开发了一种相关的方法,利用单个一维嵌入的紧凑性,以热图形式来探索成百上千个基因的表达模式。这种方法还可以发现新的标记基因,并根据它们沿着细胞的一维t-SNE平滑表达模式来组织基因。用户可以根据热图中数据集的聚类结构,来放大并识别表征和区分嵌入不同区域的基因(图2)。
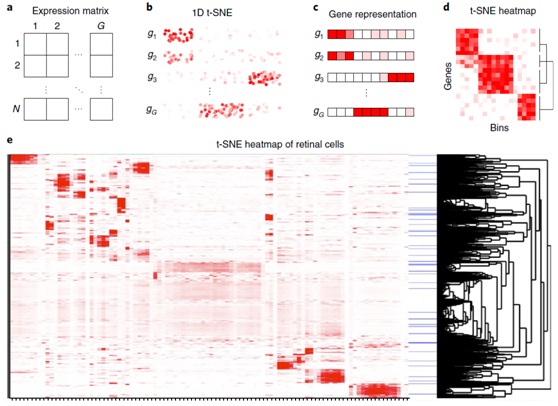
图2、t-SNE热图的示意图。a, b)从表达矩阵开始,计算1D t-SNE,转换为由每个基因的表达着色的水平轴。c)将1D t-SNE进行bin处理,通过每个bin中每个基因的平均表达来表示每个基因。d)生成vector的热图,从而将t-SNE中具有相似表达模式的基因分组在一起。e)利用已报告视网膜双极细胞数据展示t-SNE热图效果图。
关于天昊
天昊生物具有多年基因组、转录组和表观组检测与分析经验,现推出的10x单细胞转录组测序可为您提供专业便捷的科研服务及个性化的单细胞信息挖掘,期待成为您单细胞测序分析的优质服务提供商!
欢迎联系我们具体咨询!
邮箱:techsupport@geneskies.com
电话:400-065-6886