在不同条件下获得的生物过程的时间序列转录组对于鉴别过程的调控因子及其调控网络非常有用。然而,这样的数据是三维(3D)的(基因表达、时间和条件),目前没有任何方法可以处理它们的全部复杂性。这里,研究者开发了一种不同条件下无需时间点一致及均一化的方法。研究者应用它分析了在明暗循环和完全黑暗条件下玉米叶片发育的时间序列转录组,获得了八个时间序列基因共表达网络(time-ordered gene coexpression networks,TO-GCNs),这可以用来预测GCNs中任何基因的上游调节因子。八个TO-GCNs中的一个是不依赖光的,并且可能包括所有参与Kranz解剖学发育的基因,Kranz解剖学是C4植物高效光合作用的关键结构。使用这个TO-GCN,本文预测并实验验证了一个位于SHORTROOT1上游的Kranz解剖学关键调节子。此外,应用该方法还比较了玉米和水稻叶片转录本,并鉴定了玉米C4酶基因和RUBISCO SMALL SUBUNIT2的调节因子。本研究不仅提供了一种强有力的研究方法,也对Kranz解剖学发育和C4光合作用的调控网络提供了新的见解。

实验结果:
LD或TD下玉米叶片的早期发育
为了说明时间序列转录组数据新方法的必要性,研究者选择研究玉米幼苗在自然光照-黑暗周期(light–dark cycle, LD, 13h光照/11h黑暗)及完全黑暗(total darkness, TD)。LD下,幼苗经历光形态建成。相比之下,在TD下,幼苗则会发生暗形态建成。众所周知,在LD和TD下生长的玉米幼苗都发育具有Kranz特征,因此LD和TD数据的比较有助于去除大量不参与Kranz解剖学发育的光调节基因。
实验所用玉米叶片转录组数据
在这项研究中,研究者在TD下获得了一组12个发育胚胎叶片的时间进程转录组(从干种子开始每6h取样测定,从0h到吸胀后72h)。通过结合前人2013年在LD相同时间点取样测定结果,并且在T00 (干种子) 共享转录组数据,最终在LD和TD下拥有两组13个转录组。序列reads经过处理和比对,以及RPKMs的标准化。如果在25个转录组数据中至少有2个转录组的RPKM>1,就被认为是一个表达的基因。总共有25489个基因,包括1718个TF (转录因子)基因被认定是表达的,并用于后续分析。
3D转录组数据分析的方法学探讨
当比较从两个不同的实验项目中获得的两组转录组时,考虑时移效应是很重要的。然而,这并不简单,因为基因之间的时移程度不同。为了克服这一点,本研究首先考虑在每组时间序列转录组基因的共表达,然后比较LD和TD下的共表达模式(图1A )。具体来说,研究者考虑两个在LD下共表达的基因是否也在TD下共表达,反之亦然。然后利用基因共表达关系构建GCNs。此外,由于扮演就利用的数据是时间进程数据,因此方法还可以确定GCN中节点的时间顺序(图1B )。时序GCN可以揭示基因功能的动态和生物过程的时间转变(图1B )。下面,具体解释如何计算基因共表达关系,然后构建TO-GCNs。
基因共表达关系和网络计算
为了简单起见,研究者首先关注TF基因。本研究定义了两个TF基因之间的共表达关系,或者TF基因和非TF基因之间的共表达关系,如下所示。首先,分别计算LD和TD下1718个表达的TF基因的所有对的原始RPKM值的Pearson相关系数(PCC),发现任何两个TF基因之间的PCC超过0.84的概率为P<0.05。对于TF和非TF基因之间的共表达,阈值是相似的。因此,如果PCC≥0.84,将两个基因定义为正共表达(根据数据集表示为LD+或TD+)。此外,如果-0.5≤PCC<0.5,即将两个基因定义为非共表达(表示LD0或TD0),如果PCC<-0.5,研究者将两个基因定义为负共表达(表示LD-或TD-)。综合考虑LD和TD下的共表达状态,认为如果两个基因在LD和TD下都是正共表达的,那么它们属于LD+TD+关系的集合。类似地,如果它们只在LD (或TD )下正共表达,两个基因属于LD+TD0 (或LD0TD+ ),而在TD (或LD )下不正共表达。研究者对非表达基因( LD0TD0 )不感兴趣。只检测了LD+TD+,这可能包括Kranz解剖发育中的所有关键基因。
在LD+TD+集合中,共表达关系依赖于光效应,这使得研究者能够缩小Kranz解剖结构的细胞调节因子。LD+TD+中的1275个TF基因中,1207个形成了一个大的、主要的TF GCN,TF基因的节点通过共表达关系连接起来。这个GCN被称为光不依赖的TF GCN。剩余的68个TF基因形成28个GCNs,每个GCNs具有<10个TF基因。由于GCN中连接的TF基因在时间进程中具有相似的上调或下调时间点,研究者可以推断主要GCN中所有TF基因在叶片发育期间的表达时间顺序。为此,研究者选择ZmARF2-1 (AUXIN RESPONSE FACTOR2-1;Zm00001d041056)作为初始节点,因为它是LD下T00处具有峰值表达的第一个共表达模块,也就是说,它在时间0处的表达水平非常高(RPKM 96),并且单调下降,直到T72。此外,ZmARF2-1是生长素反应因子,生长素是细胞分裂和幼苗发育中重要的植物激素。这些观察结果与假设相符,即ZmARF2-1在萌发过程中起作用。然后,应用广度优先搜索(BFS)算法为所有TF基因分配时间顺序级别中的主要GCN。将这个主要的GCN称为与光无关的TF TO-GCN,它由15个时间顺序级别组成(在图2A中表示为L1至L15)。
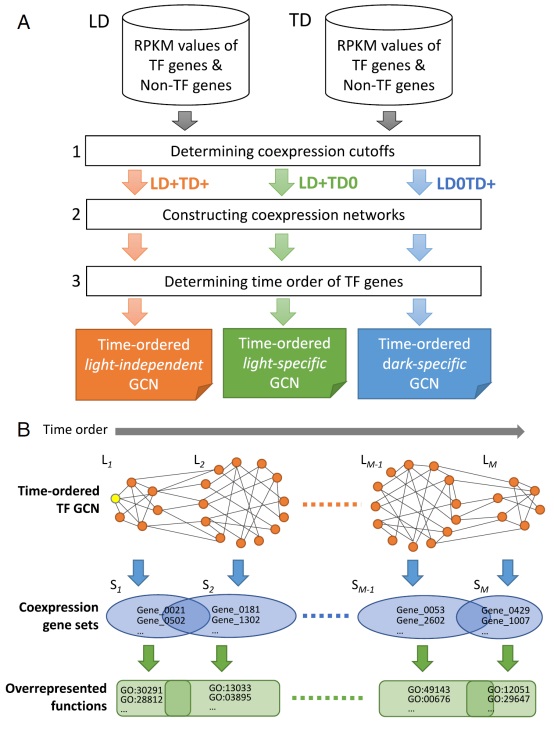
图1、比较转录组学方法流程图。(A)构建转录因子(TF)时序基因共表达网络的三个步骤。以独立于光(LD+TD+)、暗特异性(LD0TD+)和光特异性( LD+TD0) GCNs为例显示。+/-,正/负共表达。(B) 在TO-GCN中代表时间顺序中TF基因上调的水平(L1至LM),对应于不同水平的共表达基因集(S1至SM) (包括非TF基因)以及过度表达功能。L1中的黄色节点代表初始节点。一个集合中的基因可能在多个水平上与TF共同表达,因此它们可能属于多个集合。
独立于光的TF TO-GCN分析
如上所述,独立于光的TF TO-GCN中的TF基因被分配到15个水平(图2A)。这些指定的水平与LD和TD下13个时间点上TF基因的表达时间顺序相匹配,如平均标准化RPKMs (z scores)的两个热图中沿对角线的红色正方形(高表达水平)所示(图2B和C)。此外,高表达时间周期在连续水平之间重叠,表明某一水平的TF基因可能是下一水平TF基因的调控者。此外,相同水平的大多数TF基因在TD下比LD下更早被上调或下调(图2B和C )。例如,L1的TF基因在LD下的T12下调,而在TD下的T06下调(图2B和C )。
独立于光的功能分析
通过在与光无关的TO-GCN (图2A和数据集S2)的每个水平上识别共表达基因中过度表达的功能类别,研究者发现L8和L9之间有明显的发育阶段转变(图2D )。在最初的八个水平上,泛素化介导的蛋白质降解比例过高。在前四个水平上,其他九个功能也过度表达,包括,例如,蛋白质翻译后修饰、脱落酸的激素代谢(ABA)、生物和非生物胁迫、线粒体膜上的代谢物转运体和钾的转运(图2D)。
植物激素是引发发育重编程的关键。研究者发现与ABA信号转导相关的基因往往在前三个水平出现,但随后会减少,这与ABA维持种子休眠直至萌发开始一致(图2E )。与ABA相反,大多数与赤霉素(GA)信号转导相关的基因出现在L2和L3,这与GA对ABA诱导萌发过程的拮抗作用一致。大多数与生长素和细胞分裂素(CK)信号转导相关的基因分别出现在L9至L15和L10至L13 (图2E)。这也与以前的研究一致,即这两种激素一起在调节叶细胞增殖和分化中起主要作用。
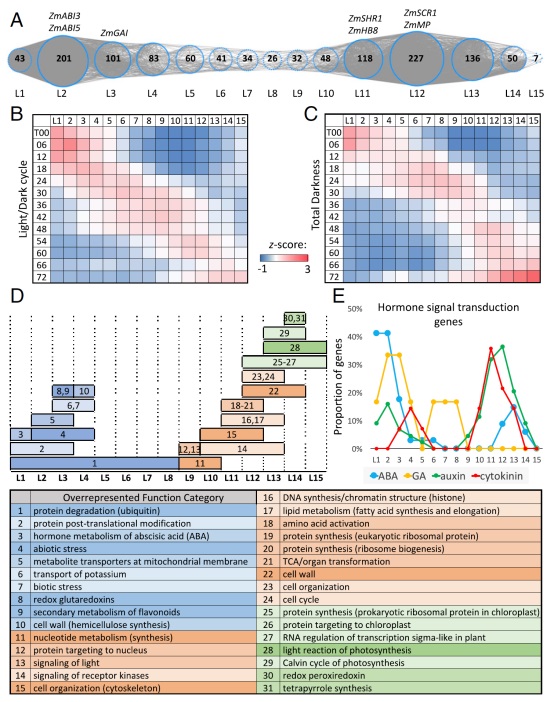
图2、与光无关的TF TO-GCN和不同水平TF基因的标准化基因表达谱。(A)以TF基因为节点的TO-GCN结构(蓝色虚线圆圈)。圆圈中的数字表示该水平的TF基因数。(B和C)分别是LD和TD下TO-GCN每个水平上TF基因每个时间点的平均标准化RPKMs (z scores)的热图。对于每个TF基因,时间点上的RPKMs首先被标准化为z scores。对于每个级别,所有TF的z scores在热图中为平均值。(D)每个水平上共表达基因的过度表达MapMan功能及列表。图中的数字(顶部)对应于表(底部)中列出的过度表示功能的索引号。蓝色、橙色和绿色分别代表发芽、叶子发育和光合作用阶段。(E)在每个TO-GCN水平上,四种激素信号转导途径中的基因比例。
关键Kranz解剖调节子的上游调节因子
ZmSHR1在玉米Kranz解剖结构的BS细胞发育中发挥了重要作用,但其上游调节因子仍未知。因此,本研究选择ZmSHR1作为例子,来展示该方法如何被用来识别特定基因的上游调节因子。具体来说,使用独立于光的TF TO-GCN设计了一个三步工作流程(图3)来识别调节ZmSHR1表达的上游调控途径。
首先,使用TO-GCN来预测ZmSHR1的候选直接调节子,该调节子应该与ZmSHR1在与ZmSHR1相同的水平上或在ZmSHR1之前的一个水平上共表达(即,图3A中的L11和L10)。(一个TF可以是同一水平上另一个TF的上游调节因子,称这些TF为一级候选调节子。类似地,研究者分别推断L9、L8和L7处的二阶、三阶和四阶候选调节子(图3A )。其次,对于每个具有未知TFBS (转录因子结合位点)的候选调节子,采用前人方法和拟南芥TFs的TFBS数据库预测其TFBS, (由图3A中的红色表示预测的TFBSs)。对于那些在图3A中用橙色表示的TFs,我们无法预测它们的TFBSs,因为玉米和拟南芥之间的DNA结合域太大,或者没有拟南芥数据。第三,对于具有已知或预测TFBS的每个TF,检查了每个可诱导靶基因启动子区域中TFBS的存在。该过程进一步将图3A中的网络简化为图3B中的网络。
接下来,我们使用电泳迁移率转移分析(EMSA)和原生质体瞬时转化分析(PTA)来验证对候选调节因子及其假设目标基因的预测。EMSA和PTA实验都支持由图3B中的红色箭头表示的上游调节级联关系;也就是说,ZmARF1-2是ZmWRKY39的直接上游调节子,而ZmWRKY39又是ZmMYB117的直接上游调节子,然后ZmWRKY39充当ZmWRKY39的直接上游调节子。
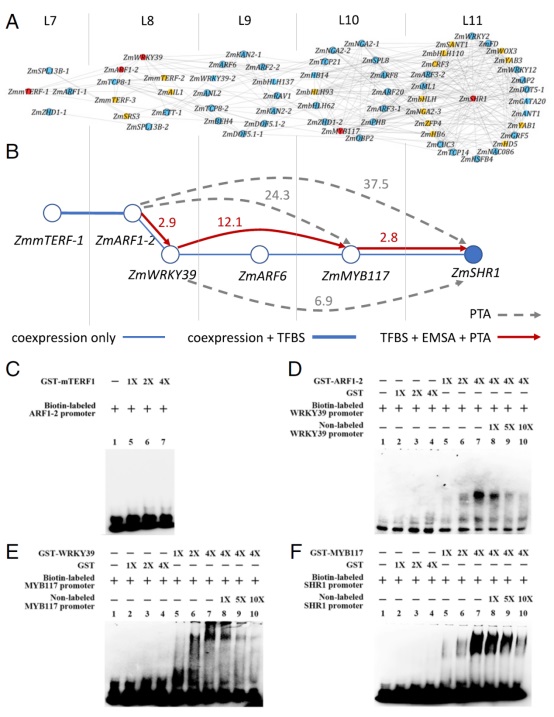
图3、玉米叶片发育中ZmSHR1基因候选上游调节因子的推断和实验验证。(A)从独立于光的TF TO-GCN推断出的ZmSHR1的一阶至四阶候选上游调节因子。ZmSHR1位于第11层子网的中心。没有已知转录因子结合位点(transcription factor binding site,TFBS)的TFs以橙色显示。已知的TFs TFBSs (蓝色)用于检测它们的候选下游基因启动子序列中是否存在它们的定位位点。TFBS支持的预测调节途径中的TFs以红色显示,并用于实验验证。(B)针对实验验证的ZmSHR1上游调控网络。实线方向或带箭头的虚线旁边的数字代表原生质体瞬时转化实验(protoplast transient assay,PTA)实验中潜在直接或间接靶基因表达水平的倍数增加。红线:目标基因启动子中TFBS的存在(如箭头所示),并经PTA和EMSA验证。灰色虚线:支持PTA,但在启动子中没有发现TFBS。(C-F) EMSA实验,测试推定靶启动子中预测的TFBSs与GST-ZmmTERF1 (C)、GST-ZmARF1-2 (D)、GST-ZmWRKY39 (E)或GST-ZmMYB117 (F)之间的相互作用。在每种情况下:泳道1:单独生物素标记的探针;泳道2至4:随着纯化GST量的增加(C除外);泳道5至7:随着GST-TF量的增加;和泳道8至10:随着未标记探针数量的增加(C除外)。
鉴定C4酶基因的调节子
为了展示我们方法的广泛应用,研究者分析了Wang等人的3D数据。这是分别来自玉米(C4)和水稻(C3)第三发育叶片的15和11段的两组转录组。请注意,这里我们考虑的是空间点,而不是时间点。按照本研究方法,研究者成功找到并鉴定了那些关键C4酶基因共表达的TF基因,并且候选TF通过EMSA实验得以验证。
结论:
这项研究的主要贡献是比较不同条件(或组织/器官或物种)和时间点(或空间点)下获得的3D转录组的方法,展示了一种分析3D数据集的有效方法的实用性。它可以在广泛的背景下应用于对比基因共表达谱。