摘要:天昊客户发表绵羊遗传育种文章
由兰州大学草地农业科技学院乐祥鹏老师课题组与甘肃农业大学动物科学技术学院、
青海大学农牧学院、甘肃省肉羊繁育生物技术工程实验室等单位共同合作的研究成果近期发表在动物遗传育种著名期刊《Animals》上,研究成果发现候选基因中有九个单核苷酸多态性与湖羊或小尾寒羊的窝产仔数显著相关,可以作为有用的遗传标记来改善窝产仔数的选择。在这项研究中使用了天昊生物SNPscan®高通量SNP分型技术。在恭喜客户发表文章同时,我们也跟大家分享一下文章的研究思路。

英文题目:Association of Polymorphisms in Candidate Genes with the Litter Size in Two Sheep Breeds
中文题目:两个绵羊品种候选基因多态性与产仔数的关系
期刊名:Animals
影响因子: 1.832
发表时间:2019-11-12
研究摘要:
湖羊和小尾寒羊是中国饲养最广泛和最著名的母羊品种,以早熟、常年发情和高繁殖力(每胎1-6只羔羊)著称。因此,增加这两个品种的产仔数对集约化养羊业至关重要。这项研究的目的是确定与绵羊产仔数相关的潜在遗传标记,这些标记位于10个基因上。本研究采集了537只湖羊和420只一胎仔数小尾寒羊的血样。湖羊和小尾寒羊的平均产仔数分别为2.21和1.93。用DNA混池测序法检测了与卵泡发育和雌性生殖相关的10个基因中潜在的单核苷酸多态性。SNPscan®用于单独基因分型。本实验在10个候选基因中的九个(除了NOG)中78个可能的SNPs进行了检测,共成功地鉴定出50个SNPs。质量控制后,湖羊42个SNPs和小尾寒羊44个SNPs最终用于进一步分析。关联分析显示6个基因中有9个SNPs与湖羊或小尾寒羊的产仔数显著相关。两个位点(LIFR: g.35862868C>T 和 LIFR: g.35862947G>T)单倍型的组合分析表明,在小尾寒羊中H2H3 (CTTT)单倍型组合产仔数比其余单倍型组合更多,也比单独突变的多。总之,本研究表明,6个基因中的9个重要的SNPs可以作为绵羊标记辅助选择的有用遗传标记。
研究背景:
产仔数是最重要的经济特征之一,它对养羊业的盈利能力有显著影响。如果确定了与产仔数相关的特定遗传标记,那么标记辅助选择可以用来改进产仔数的选择。基因组选择是有效的,但是在大样本的情况下是非常昂贵的。在候选基因中发现与产仔数相关的单核苷酸多态性(SNPs)是在预算有限的情况下改善产仔数的另一种方法。众所周知,排卵率和胚胎存活与绵羊产仔数直接相关。因此,扫描这些具有已知生殖生理功能的基因中的SNPs是一种有效的方法。根据现有文献,选择动物中具有已知生殖生理功能的10个基因(NGF、NTRK1、KIT、KITLG、LIF、LIFR、NCOA1、ADAMTS1、NPM1和NOG)作为绵羊产仔数的潜在候选基因(图1)。
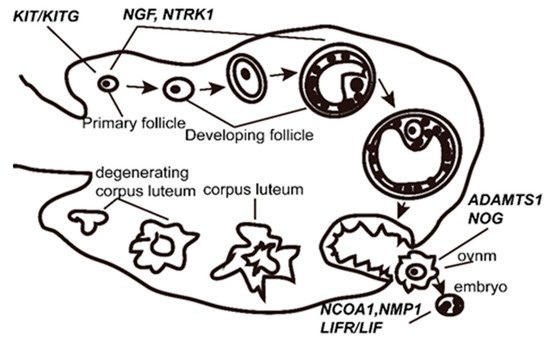
图1、十个候选基因的生殖生理功能
在中国,2017年家养绵羊的数量为1.1635亿只,绵羊肉占反刍动物肉类产量的14.38%。湖羊和小尾寒羊是两大绵羊品种,在我国广泛用于集约化养羊系统(舍饲)。湖羊和小尾寒羊的估计库存分别约为400万和500万只。这两个品种通常作为雌性亲本与优良公羊品种杂交来生产肉类。
尽管这10个基因在影响生殖生物学过程中的作用有相对明确的特征,但是对这些用于绵羊产仔数遗传选择的候选基因的评估还没有广泛和系统地进行,特别是在湖羊和小尾寒羊中。本研究假设上述10个候选基因中的潜在SNPs可能与湖羊和小尾寒羊的产仔数有关。因此,本研究的目的是扫描这10个基因中的SNPs,并研究湖羊(n = 537)和小尾寒羊(n = 420)的SNPs与产仔数的关系。本研究的发现可能作为湖羊和小尾寒羊的有用遗传标记。 材料和方法:
表型数据收集和DNA提取:
在本研究中,所有母羊(湖羊和小尾寒羊)在相同的管理条件下饲养,并于2014年9月至12月交配,2015年2月至4月,采集了957只母羊血样并记录已产仔数,其中湖羊537只,小尾寒羊420只。本实验流程如图2所示,湖羊和小尾寒羊产仔数记录表型数据如图3所示。湖羊的产仔数从1到5只不等,小尾寒羊从1到4只不等。湖羊和小尾寒羊的平均产仔数分别为2.21和1.93。基因组DNA用苯酚-氯仿法提取,然后溶解在双蒸水中,在-20℃储存。
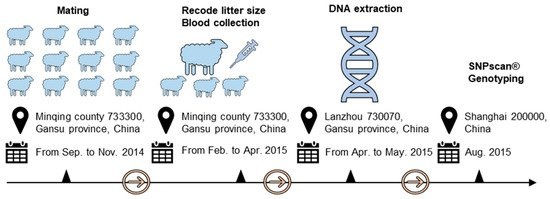
图2、实验方案示意图
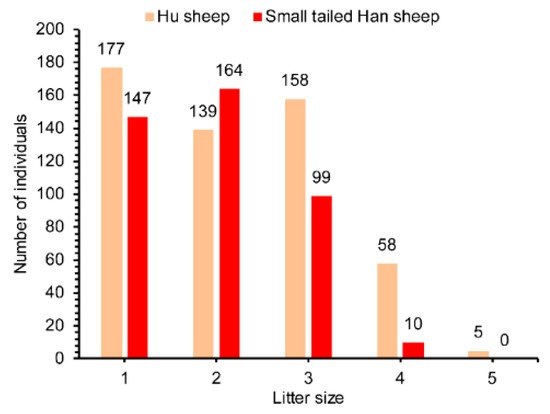
图3、湖羊和小尾寒羊产仔数的频率分布
SNP检测和基因分型
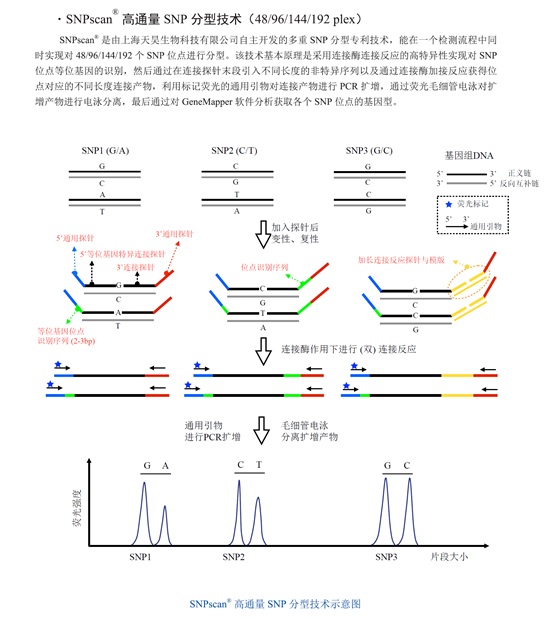
总共为两个绵羊品种构建了10个DNA池,每个DNA池由10只个体组成,以鉴定所分析的10个基因中的潜在SNPs。如表1所述,这些个体是在所有不同的产仔数之间随机选择。总共设计了119对引物来扩增每个基因的所有外显子和侧翼区域,对PCR产物进行测序,通过对序列进行比较来检测可能的SNPs。所鉴定的SNPs通过天昊生物SNPscan®高通量SNP分型技术分别进行基因分型,该方法基于连接酶连接和多重荧光PCR反应进行。从957个样品中随机选择三个DNA样品进行两次基因型鉴定,两个空白样品(ddH2O)用于消除交叉污染。
表1、每个DNA池中的样本构成在不同的产仔数之间维持平衡

群体参数计算与数据质量控制
利用SnpReady R包用于计算等位基因频率、次要等位基因频率(MAF)、多态信息含量(PIC)、预期杂合性(He)和观察杂合性(Ho),并进行Hardy–Weinberg平衡检验。如果MAF < 0.05和/或严重偏离哈迪-温伯格平衡(p < 0.001),则对这些SNPs进行排除不再进行进一步分析。
SNP关联分析
使用线性模型来对单个SNP和每个绵羊品种产仔数的影响进行检验:L = 1n m + G + e
,其中L是产仔数的n × 1向量(湖羊,n = 537小尾寒羊,n = 420),1n是所有元素等于1的n × 1向量,μ是总体平均值,G是对应于SNPs的固定效应,e是随机剩余效应的n × 1向量。基因型间平均产仔数的差异用R包中的LSD最小二乘法检验。P < 0.05被认为是显著的。P < 0.01被认为是非常显著的。Bonferroni校正用于基因型组之间的多重测试。
单体型分析
在目前的研究中,只有每个绵羊品种同一基因中显著关联的SNPs被用来估计连锁不平衡和单体型区块的程度。湖羊的两个显著的SNPs和小尾寒羊的四个显著的SNPs满足这一前提,并且使用Haploview 4.2软件估计显著SNPs对和单体型区块之间的LD程度。
研究结果:
基因分型和数据质量控制
通过DNA混池测序,在9个候选基因(除了NOG)中共鉴定出78个潜在的SNPs。
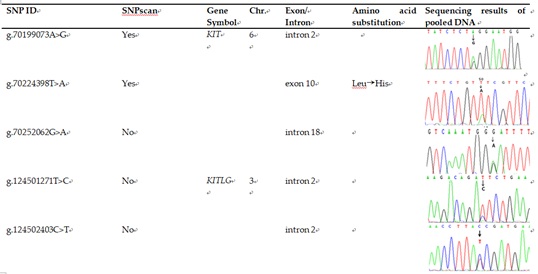
补充材料、10个候选基因中78个鉴定SNPs的信息(部分)
在评估引物后,78个单核苷酸多态性中的50个使用SNPs扫描方法进行了基因分型。在两个空白样品中检测到三对技术重复的基因型相关系数等于1且没有基因型信号,表明SNPscan®高通量SNP分型技术方法是可靠的。排除MAF < 0.05和/或严重偏离哈迪-温伯格平衡(p < 0.001)的8个湖羊SNPs和6个小尾寒羊SNPs,最后总共42个湖羊SNPs和44个小尾寒羊SNPs被用于进一步分析(表2)。
表2、湖羊(HUS)42个SNPs和小尾寒羊(STH)44个SNPs的群体遗传参数表(部分)
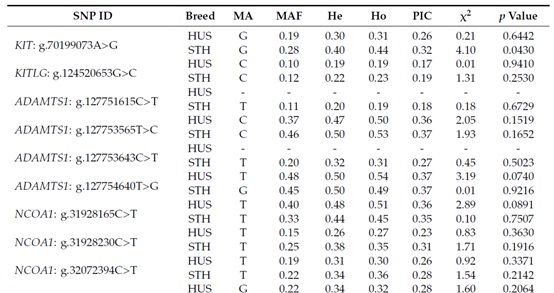
注:MA:次要等位基因;MAF:次要等位基因频率;PIC:多态信息内容;He:预期杂合性;Ho:观察杂合性;x2和p值表示哈迪-温伯格平衡检验的卡方值和p值;“-”表示信息不可用。
与产仔数相关的SNPs
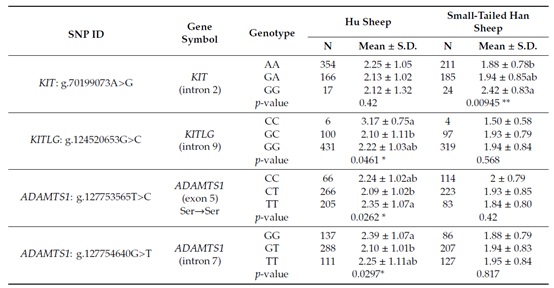
表3详细说明了检测的SNPs和产仔数之间的显著关联性。单个SNP关联分析表明,湖羊的3个SNPs及小尾寒羊的4个SNPs是与产仔数显著相关的,另外小尾寒羊中有2个SNPs是与产仔数为极显著相关。其余36个SNPs与产仔数无显著相关性。
表3、湖羊和小尾寒羊9个SNPs与产仔数的关系(部分)
.
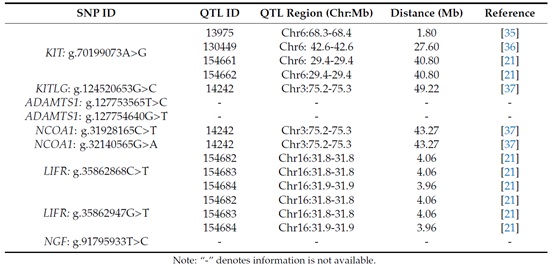
9个重要的SNPs被映射到绵羊QTL数据库(https://www.animalgenome.org/cgi-bin/QTLdb/OA/index),以更好地理解这些SNPs的潜在作用。9个SNPs被定位到13个与产仔数相关的数量性状位点(表4)。SNPs和QTL之间的最近距离为1.8 Mb。
表4、湖羊和小尾寒羊与产仔数相关的9个显著相关SNPs的数量性状位点注释
单体型分析
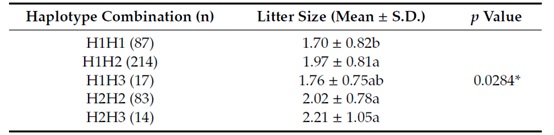
D′(r2)值是LD的一个指标,在本研究中进行了计算。D′(r2)分别为0.72 (0.34)和0.30 (0.04),表明湖羊有2个SNPs (ADAMTS1: g.127753565T>C和ADAMTS1:g.127754640T>G) (图4A)和小尾寒羊有2个SNPs (NCOA1: g.31928165C>T和NCOA1:g.32140565G>A)(图4B)都并不紧密连锁。而小尾寒羊中的2个SNPs (LIFR:g.35862868C>T和LIFR: g.35862947G>T))是紧密连锁的(图4C,D′和r2分别为0.97和0.84)。在小尾寒羊中鉴定出一个单倍型区块(图4C),其中四个不同的单倍型是H1 (CG)、H2 (TT)、H3 (CT)和H4 (TG)。单体型频率最高的是H1,占所有单体型的48.7%。H2是第二大比例,为47.1%,紧随其后的是H3 (3.7%)和H4 (0.5%)(图5)。由于低频率的单倍型在统计分析中没有意义,H4被排除在后续分析之外。关联分析表明,单体型组合与小尾寒羊的产仔数显著相关(表5)。H2H3 (CTTT)、H2H2 (TTTT)、H1H2(CTGT)个体的产仔数大于H1H1 (CCGG)个体。
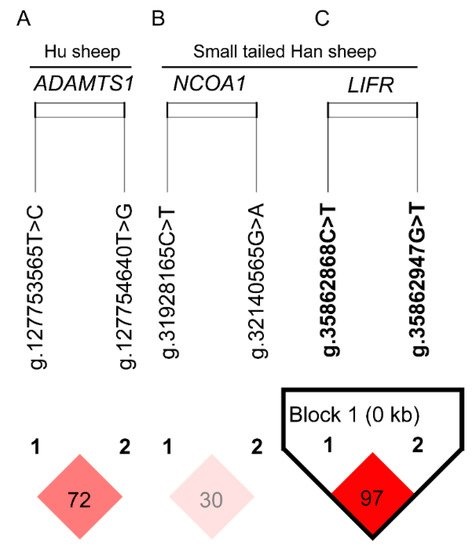
图4、ADAMTS1 (A)、NCOA1 (B)和LIFR (C)基因中显著性SNPs的连锁不平衡模式图。红色表示连锁不平衡程度(D’值)
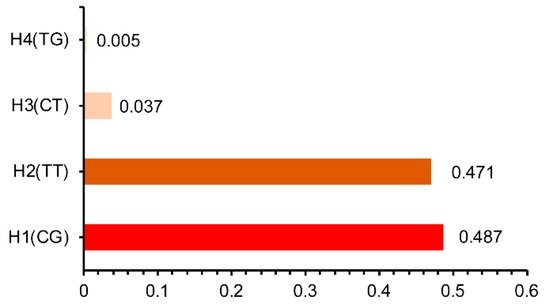
图5、小尾寒羊两个基因座(LIFR: g.35862868C>T和 LIFR:g.35862947G>T)的单倍型频率
表5、小尾寒羊单体型组合与产仔数的关系表
结论:
本研究系统地研究了10个基因中潜在的SNPs作为绵羊产仔数增加的候选标记。对6个基因中的9个SNPs (KIT:g.70199073A>G, KITLG: g.124520653G>C, ADAMTS1: g.127753565T>C, ADAMTS1: g.127754640G>T, NCOA1: g.31928165C>T, NCOA1: g.32140565G>A, LIFR: g.35862868C>T, LIFR: g.35862947G>T 和NGF: g.91795933T>C)和2个基因座单倍型分析表明,H2H3 (CTTT)组合单倍型在小尾寒羊中产仔数比其他组合单倍型多,这9个SNPs可作为小尾寒羊和湖羊群体遗传标记。
昊技术:
关于天昊
截止到2019年1月,天昊生物的SNP分型平台共发表文章366篇,总影响因子1057分。除了SNP分型外,我们还提供动植物拷贝数变异(CNV)检测服务,欢迎联系我们具体咨询!
邮箱:techsupport@geneskies.com
电话:400-065-6886