染色质可及性(Chromatin accessibility)是指核大分子能够与染色质化的DNA进行物理接触的程度,由核小体及其他阻碍接近DNA的染色质结合因子的占据和拓扑结构所决定。核小体是染色质的核心结构元件,由一个组蛋白八聚体组成,周围环绕着约147 bp的DNA。核小体的组成和翻译后修饰反映了不同的功能状态,并通过多种机制调节染色质的可及性,如通过空间位阻改变转录因子(TF)结合,调节核小体对活性染色质重塑因子(remodellers)的亲和力。基因组中核小体的拓扑结构是不一致的:虽然在兼性和组成性异染色质内密集排列,但组蛋白在调节位点(包括增强子、绝缘子和转录的基因体内)是缺失的。核小体间DNA常与TFs、RNA聚合酶或具有连接组蛋白(linker histones)的结构蛋白结合,这有助于高级染色质的结构并调控对DNA的接近。核小体占据和连接组蛋白占据在基因组范围内呈动态变化,构建了一个从封闭染色质到高度动态,可接近或开放染色质的可及性连续体 (图1)。染色质可及性图谱广泛反映了调控的能力,并不是一个静态的生物物理状态,其对染色质的结构和功能至关重要。
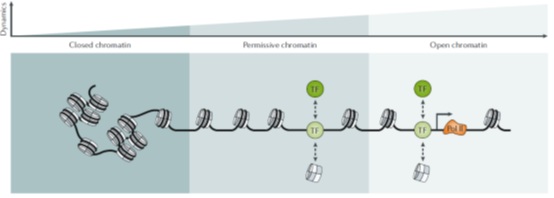
图1 可及性状态反映了染色质动态变化过程
可接近的基因组虽然仅占总DNA序列的2~3%,但却捕获了90%以上的TFs结合区域(ENCODE数据)。除了少数在兼性或组成性异染色质中富集的TFs外,在ENCODE项目中研究的绝大多数TFs几乎特异性地与开放染色质结合。TFs与组蛋白和其他染色质结合蛋白进行动态竞争,调节核小体占据并促进局部接近DNA;反过来,特定细胞类型的可及性图谱也可以调控TF结合。对于多细胞系统,TFs具有广泛的功能作用,在短时间尺度上提供转录的动态调控,并建立和维持具有共同基因组的细胞类型的持续表观遗传渠道化(epigenetic canalization)。因此,染色质可及性既反映了整合的TF结合,也反映了遗传位点的调控潜力。这一观点为追踪决定细胞状态的转录调节因子差异结合的可及性变化奠定了分析基础。
染色质可及性普遍通过量化染色质对酶甲基化或其组成DNA切割的敏感性来检测(图2)。原则上,染色质可及性的检测取决于所研究的分子;然而,据报道可及性又具有显著的保守性。1973年,Hewish和同事使用DNA内切酶来片段化染色质,表明核小体在整个基因组中具有周期性的超敏反应。这种周期性可以通过Southern blot杂交检测,发现在基因组保守的DNA酶超敏位点(DHSs)之间存在一个典型的100-200bp相位模式。这和随后的工作为典型的核小体相位提供了最早的直接证据。类似的技术也被用于将黑腹果蝇染色质重塑与热休克位点的同时转录激活联系起来。随着1985年PCR技术的引入,各种定量方法(如下所述)已经发展起来,可以使用核酸内切酶和连接介导的PCR技术来检测位点特异性的染色质可及性。
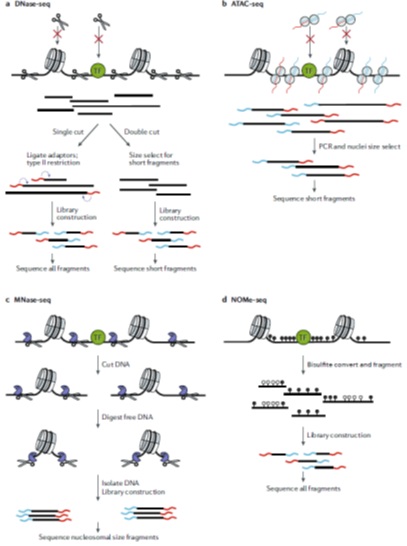
图2 染色质可及性检测方法原理
DNase-seq
全基因组范围内的开放染色质区域检测首次在2006年的两篇研究中发表,研究通过将DNase I消化后的天然构象的染色质片段杂交到覆盖1%人类基因组的平铺芯片上得到这部分序列信息。后续改进的技术流程采用短读长测序来定量基因组范围内DNase敏感的染色质相对丰度(DNase I超敏位点测序(DNase- seq)) (图2a)。Boyle等人使用II型限制性内切酶分离并对每个DNA酶切位点进行条形码标记(单切,single cut),而Hesselberth等人使用严格的片段选择来富集DHSs中成对切割事件产生的可测序片段(双切,double cut) (图2a)。尽管这些测序方法之间有广泛的共识,Boyle的实验方法可以鉴定到更多可接近的位置,而Hesselberth方法则提供了一个简化的实验流程,并且其中捕获的来自广泛的不可接近染色质的片段会更少(更高的信噪比)。总的来说,这些基因组水平的染色质可及性检测显示,少数DHSs位于启动子和转录起始位点(TSS)的近端区域,超过80%的可接近性区域位于远端增强子区。
ATAC-seq
转座酶可接近染色质测序(ATAC-seq)使用超活性Tn5转座酶将Illumina测序接头插入可接近的染色质区域(图2b)。与双切DNase-seq操作方法类似,ATAC-seq选择性地放大可接近染色质的近端双切事件。ATAC-seq可及性检测与双切(r > 0.8)和单切(r > 0.75) DNase-seq实验结果均高度相关,尽管在做一些更高分辨率的分析时(例如分析TF印记),两种技术在测序偏倚性上仍存在差异。由于Tn5介导的接头连接的高效率,已经可以对低至500个数量的细胞成功构建高度复杂的ATAC-seq文库。近期也有报道多种ATAC-seq的改进方法,包括Sos等人的一项研究表明,可以利用单个转座事件的体外转录来构建文库。ATAC-seq之所以被广泛采用,部分原因是它能够很好地识别可接近的染色质,而且简单、快速实施,并能应用于现实条件下有限的临床和原代组织样本。值得一提的是,10000-20000个细胞的ATAC-seq文库通常在2小时内完成,相比较而言,DNase-seq通常需要数百万个细胞的多天操作流程。尽管已知两种方法的局部序列偏差不同,但ATAC-seq和DNase-seq捕获了相似的调控信息。
MNase-seq
鉴于组蛋白在调节染色质可及性方面的中心作用,核小体占据和定位技术——微球菌核酸酶测序(MNase-seq)最近被应用于检测可及性(图2c)。MNase既可作为核酸内切酶来切割核小体间DNA,也可作为外切酶来降解不受蛋白质保护的片段化DNA。MNase-seq与DNase-seq和ATAC-seq之间的一个显著区别是后两者都没有发生核小体DNA切割事件。事实上,MNase可能通过其内切酶活性来切割核小体DNA;然而,这类切割事件是否真实存在,因为其自身外切酶介导的片段降解而无从得知。由于MNase对核小体DNA的切割效率低于核小体间DNA,因此该酶被广泛用于分离跨越单个核小体的片段。先前的研究显示,某些核小体对MNase的敏感度具有剂量依赖特性,Mieczkowski和其同事利用不同浓度的核酸酶进行MNase-seq,以检测整个基因组的差异可及性。这种技术,称为MNase可及性(MACC),基于MNase-seq文库中可接近DNA随核酸酶浓度的增加而减少(可能由于外切酶介导的消化),而受保护的核小体DNA由于内切酶介导的切割发生率增加而表现为相对丰度增加。MACC信号在预测活跃调控区域(组蛋白H3赖氨酸27乙酰化(H3K27Ac)阳性区域)高度富集,与TSSs、启动子和远端增强子的DNase超敏数据相关性较好,但在转录终止位点或基因体内相关性差。这种差异可能是由于不同方法中片段长度的差异富集或使用的酶探针大小的不同造成。
NOMe-seq
核小体占据和甲基化组测序(NOMe-seq)使用来自M.CviPI的GpC甲基转移酶(MTase),这种酶通过对可接近的DNA进行化学修饰而非切割的方式来检测染色质的可及性(图2d)。这种新的MTase在GC二核苷酸上产生异常的甲基化,与人类和小鼠基因组中常见的CG二核苷酸上的内源性甲基化区别开来。因此,NOMe-seq在高分辨率下可以同时检测DNA可及性和甲基化状态,因为GpC在整个基因组中频率很高。重要的是,NOMe-seq并不是一种富集方法,因此需要相对大量的测序reads来获得足够的深度来确定整个基因组的可及性水平。然而,由于富集偏差的缺失和该技术的单分子特性,使得其对染色质可及性图谱的检测比DNase-seq、ATAC-seq或MNase-seq更能提供定量的信息,因为每个基因组位点的相对可及性水平可以直接测定。
单细胞可及性检测
单细胞可及性变化的检测有望揭示基因组调控的一个核心问题:染色质可及性短时间尺度内的波动和发展变化是如何在整个基因组中协调的?基于ATAC-seq、DNase-seq和NOMe-seq文库制备方案,已经开发了多种方法来检测单个细胞中的染色质可及性。
组合标签(Combinatorial indexing)为成千上万的单细胞ATAC-seq文库加上条形码(barcode)提供了一种完美的策略。在这种方法中,用带有独特的条形码Tn5酶对纯化的细胞核进行多重转座反应,然后在有限稀释的条件下混样并分开进行多重标签PCR反应。这种方法的原理是任意两个细胞在转座后,被后续混样和分开PCR的操作中标记上完全相同的两重标签的概率很低,因此不需要分离单个细胞。组合标签已被用于分析黑腹果蝇胚胎发育的成千上万个单细胞的可及性,建立了13种不同组织的小鼠细胞类型图谱,并研究正在发育的小鼠前脑的转录调控。
单细胞ATAC-seq也可以通过在微流控设备(Fluidigm, C1)或在单个标签的纳米孔芯片中来(Takara Bio, ICELL8)实现捕获单细胞。微流控捕获已经被用于在早期的造血过程中对成千上万个单细胞的可及性进行分析,而纳米孔芯片有望进一步将微流控方法的通量提高一个数量级。在选择使用这两个平台还是组合标签方法时需要作出权衡:微流控和纳米孔芯片平台每次实验处理的细胞会明显少于组合标签方法,但由此产生的单细胞文库复杂性至少示后者的两倍。文库复杂度的差异是一个重要的参数,因为即使是对无所不在的开放区域也只是采样了少量分散的区域(平均而言,使用C1平台的单个细胞中只观察到所有开放区域的10%),我们预计这两种方法的文库复杂度在近期都将得到改善。单细胞ATAC-seq最近已经在10X Genomics公司和Bio-Rad Laboratories公司的基于液滴的微流控平台上得到了应用。早期的报告表明,这些高通量平台将提供类似于纳米孔捕获技术的数据质量。
转座酶介导的单细胞染色质可及性分析很快出现了DNase-seq和NOMe-seq的单细胞改进版。结合甲基化、可及性和核小体相位的原理证明以及单细胞NOMe-seq分析,Pott报道了从单细胞尺度能鉴定出5.2-9.5%的DHSs 。虽然单细胞DNase-seq在目前还不容易扩展,但它可以在所有开放染色质中捕获、生成具有近100,000个独特reads的显著复杂性的单细胞可及性片段文库。尽管DHSs的reads比例略低于基于ATAC-seq的方法(单细胞DNase-seq的占23-26%,而单细胞ATAC-seq的占35-55%),但在DHSs上观察到的独特片段的数量仍然相对较高。值得注意的是,对于一小部分DHSs,在几乎所有的单细胞中都可以从这些区域中鉴定到可接近的片段;此外,来自单个细胞的典型DNase-seq文库包含来自所有DHSs的35-59%的片段(相比之下,来自单个细胞的ATAC-seq只占10%)。总的来说,这些原理证明方法为构建单细胞表观遗传学技术手段提供了重要的研究方向。
解析染色质可及性特征
最近的单分子和单细胞可及性检测表明,细胞群的可及性检测代表了不同分子状态的总体平均值(图3)。当观察潜在的、动态的、不同步的过程时,这种分子异质性自然会出现。例如,在转录位点RNA聚合酶和其他TFs一起取代TSS上游的核小体,并迁移经过启动子下游的定位良好但不稳定的核小体(图3a)。单分子NOMe-seq数据的印记分析揭示了这一过程,捕获了转录过程进行时的每个分子状态(图3b)。尽管考虑到采样效率,可及性的单细胞检测在全基因组范围内预测的调控区域发现了相似的分子异质性(图3c)。
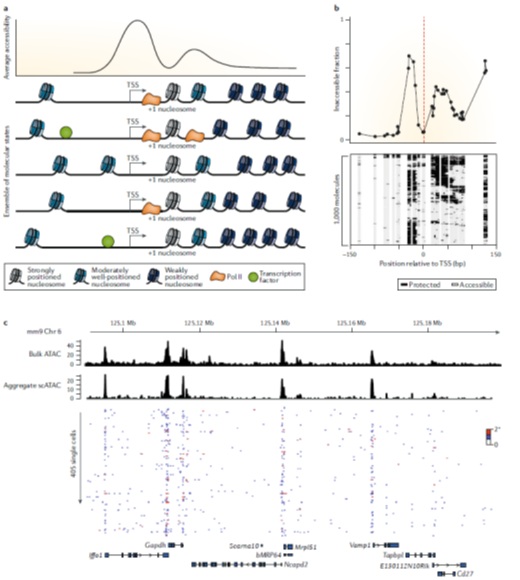
图3 细胞群范围的染色质可及性检测反映了异质性的单分子平均的可及性
DNase-seq和ATAC-seq对大量细胞群的开放染色质的检测可被视为总体的TF-结合信号的整合。结合TFs保护DNA不被酶消化,其保护程度与TF对DNA的结合占据有关。考虑到它们相对快速的脱去速率,我们可以预期,TFs通常比核小体提供更少的保护。这种精细的TF相关的保护需要使用不同的生物信息学分析方法,利用基因组范围内TF结合亲和力和酶切特异的序列偏倚性信息去解析TF结合和染色质结构。可及性结合转录数据进一步扩展了此分析流程:给定一组不同的细胞类型之间的可接近区域,在不同的发展阶段或其他实验条件下,可以发现哪些TFs是差异表达的,并被推断结合到差异的可接近的DNA上,而这些TFs可能参与调控某些表观遗传的差异。因此这仍然是一个活跃的领域研究,我们预计未来几年的焦点集中在整合可及性与其他表观遗传检测的计算方案。
最近,染色质可及性已被用来识别由全基因组关联研究(GWAS)产生的因果遗传变异。人类基因组中大多数单核苷酸多态性(SNPs)发生在非编码和基因间的基因组区域,个体对复杂性状的影响很小或是间接的,因此混淆了因果变异的检测。最近,一些研究利用染色质的可及性来更好地理解这些非编码SNPs及其对性状的影响。例如,Maurano等人发现许多因果GWAS变异集中在非编码的DHSs中,通过使用染色质可及性数据,可以更准确地关联到它们影响的基因。这些结果强调了将染色质可及性数据与功能和其他表观遗传数据分析相结合的价值。

ATAC-seq技术优势及参数
天昊生物具有多年基因组、转录组和表观组等多组学检测与分析的经验,ATAC-seq在众多领域还未曾大展身手,同时又可以联合WGS或WGBS或RNA-seq等进行多组学分析丰富您的研究内容。天昊生物可为您提供专业的科研服务及个性化的生信挖掘!
欢迎联系我们!
邮箱:techsupport@geneskies.com
电话:400-065-6886