SNP关联研究或者GWAS研究的阳性位点,大部分时候总能很优秀的落在非编码区。
这些非编码SNP到底影响了什么???
别着急,给你推荐2个数据库挖挖看,或许就有收获。

◆ENCODE数据库(Encyclopedia of DNA Elements, https://www.encodeproject.org/):由美国国家人类基因组研究所(NHGRI)在2003年发起的一个项目,主要存放了人类、小鼠、果蝇、蠕虫这4个物种的多种细胞和组织类型的各种调控过程的测序结果(The primary goal of the ENCODE project is to determine the role of the remaining component of the genome, much of which was traditionally regarded as "junk.")。这一项目的研究人员对147种细胞系进行了将近1,650次实验,涉及分子转录,转录因子结合,染色体拓扑结构,组蛋白修饰,DNA甲基化等多方面表观遗传学内容。2008年,也就是NHGRI启动ENCODE计划的五年后,NIH又开始了第二项大规模图谱绘制工程:表观基因组学路线图项目(Roadmap Epigenomics Program),这一项目整理了61个“完整”的表观基因组,并且未来还计划进行更多的研究。大量的表观遗传学数据,可能能够为非编码区的SNP调控功能提供解释。
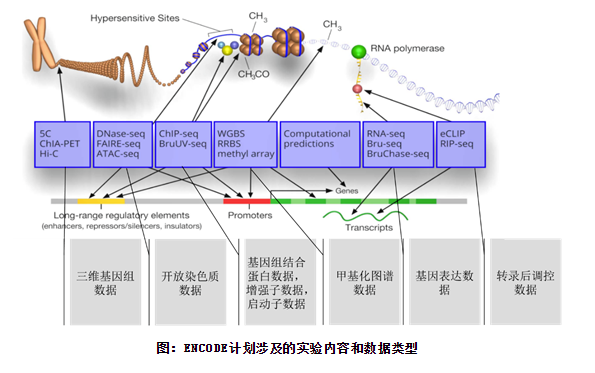
HaploReg和RegulomeDB数据库,将ENCODE数据库中的调控数据与基因组中SNP数据结合起来,从而实现了对SNP调控功能的综合评价。


●网址:
https://pubs.broadinstitute.org/mammals/haploreg/haploreg.php
●数据输入页面
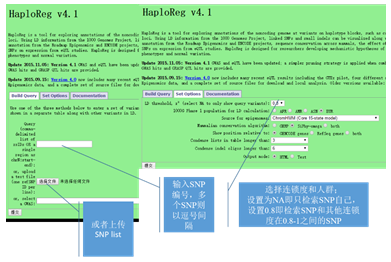
●以rs2854510为例的综合结果展示页面,每个SNP的详细注释结果需点击SNP的名字

1. SiPhy cons:序列保守性注释(SiPhy软件)。
2. Promoter histone marks,Enhancer histone markers,DNAse:来源于DNAse和ChIP-Seq实验的调控元件分类注释结果,包括是否处于组蛋白修饰位置(promoter,enhancer等);属于什么染色质状态Chromatin States(来自Roadmap项目);是否处于染色质开放区域(DHS区域)。

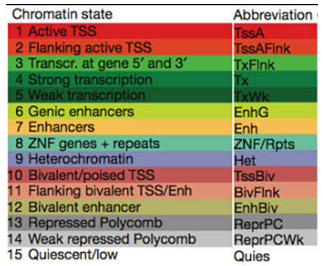
1)Chromatin states (Core 15-state model)
包括前面8个活跃状态和后面7个抑制状态,具体含义和缩写见下图。
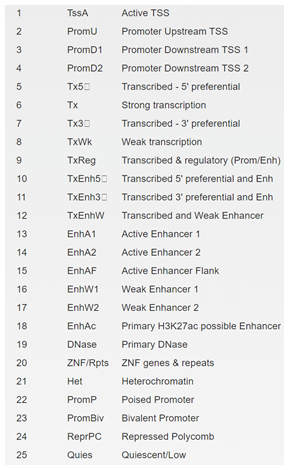
2) Chromatin states (25-state model using 12 imputed marks)
具体含义和缩写见下图。
3)H3K4me1, 与基因的转录激活有关,代表此处是转录增强子peak
4)H3K4me3, 与基因的转录激活有关,代表此处是启动子区peak
5)H3K27ac,与基因的转录激活有关,代表此处是转录增强子peak
6) H3K9ac, 与基因的转录激活有关,代表此处是启动子区peak
7) DNase,说明此处对DNaseⅠ表现出高度敏感,处于转录活跃区
3.Proteins bound :基于ChIP-Seq 实验,展示在何种细胞中SNP处于何种转录因子的结合位点.
具体信息需要查看SNP位点详细页面的信息:

4.Motifs changed:基于ENCODE 的转录因子CHIP-SEQ数据,整理了这些转录因子的基因组结合motif。这里会展示SNP的两个碱基对motif的影响。
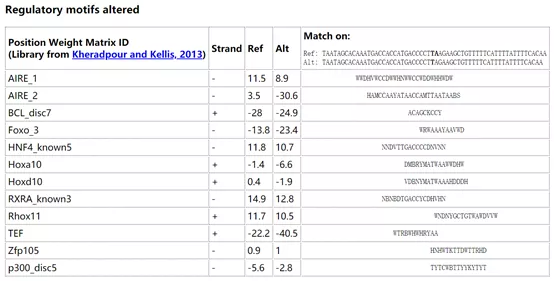
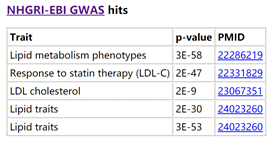
5.NHGRI/EBI GWAS hits:基于NHGRI catalog,提示该SNP是GWAS研究的阳性位点
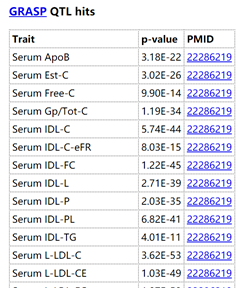
6.GRASP QTL hits:基于GRASP数据库,提示该SNP是某个性状的阳性位点
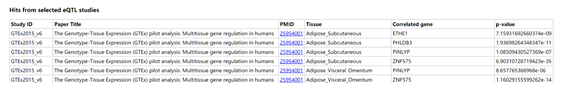
7.Selected eQTL:基于GTEx analysis V6, the GEUVADIS analysis, 和其它10篇文献,提示该SNP在XXX组织中调控XXX基因表达


●网址:
https://www.regulomedb.org/regulome-search
●数据输入页面:
●结果综合展示页面
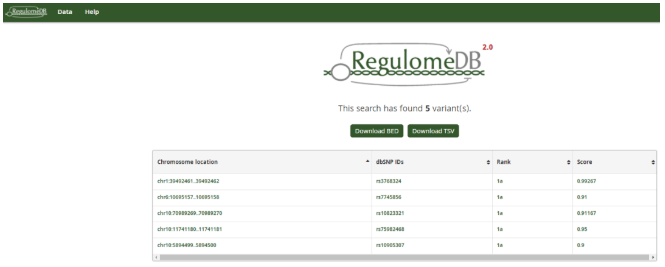
第三列Rank的解读:

第四列Score解读:
RegulomeDB概率得分从0到1不等,1代表最可能是调控变异。RegulomeDB得分基于功能基因组学特征以及实验来源的连续值,如ChIP-seq信号、DNase-seq信号、信息含量变化和DeepSEA得分等。详细信息可参考最新文献:
Dong S and Boyle AP. Predicting functional variants in enhancer and promoter elements using RegulomeDB. Human Mutation 2019, 40:1292-1298. PMID: 31228310.
概率得分和第三列的Rank之间总体上是正相关的,但也有一些例外,因为1)预测概率得分时增加了一些额外的特征数据。2)概率评分中使用的特征与rank评分的权重不同。
●点击rs3768324,获得详细注释信息
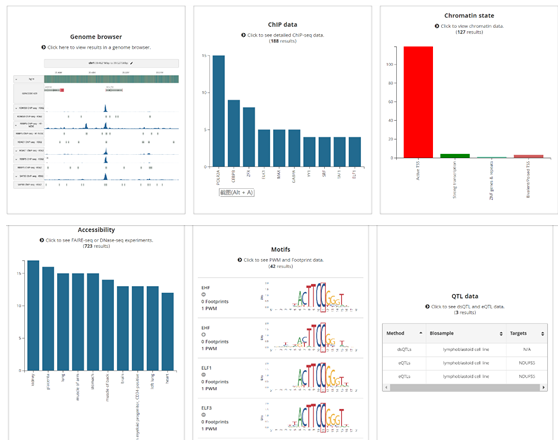
细节数据基本是和Haploreg重叠的,展示的细节略有区别,建议老师们两个数据库都进行查询和参考。

扩展阅读:
基于HaploReg和RegulomeDB数据库的生信挖掘文章:Liao, X., Lan, C., Liao, D. et al. Exploration and detection of potential regulatory variants in refractive error GWAS. Sci Rep 6, 33090 (2016). https://doi.org/10.1038/srep33090
SNP位点基于HaploReg和RegulomeDB数据库进行功能注释:Song J, Yang Y, Mauvais-Jarvis F, Wang YP, Niu T. KCNJ11, ABCC8 and TCF7L2 polymorphisms and the response to sulfonylurea treatment in patients with type 2 diabetes: a bioinformatics assessment. BMC Med Genet. 2017;18(1):64. Published 2017 Jun 6. doi:10.1186/s12881-017-0422-7
天昊生物
帮您更好的完成实验,
帮您认识更好的数据库!
欢迎联系我们!
电话:18964693703(微信同号)
公司网址:www.geneskybiotech.com
邮箱:techsupport@geneskies.com
