ggpar包是一个用于图形参数调整的包,但对于该包的使用介绍目前并不多见,这里,我们提供了官网的示例,并加以解释
(https://www.rdocumentation.org/packages/ggpubr/versions/0.1.4/topics/ggpar )。
加载R包和数据
In[1]:
.libPaths("C:/Program Files/R/R-3.6.1/library")
library(ggpubr)
In [2]:
# Load data
data("ToothGrowth")
df <- ToothGrowth
head(df)
Out[2]:
len supp dose 4.2 VC 0.5 11.5 VC 0.5 7.3 VC 0.5 5.8 VC 0.5 6.4 VC 0.5 10.0 VC 0.5

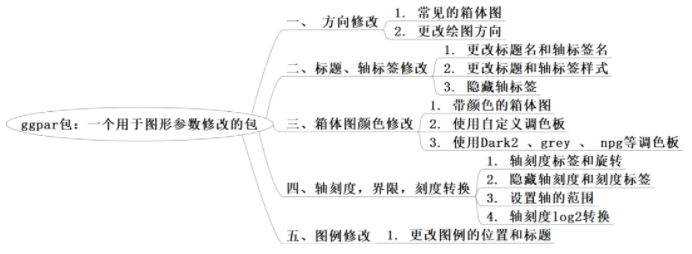
1. 常见的箱体图
In[3]:
p <- ggboxplot(df, x = "dose", y = "len") pOut[3]:
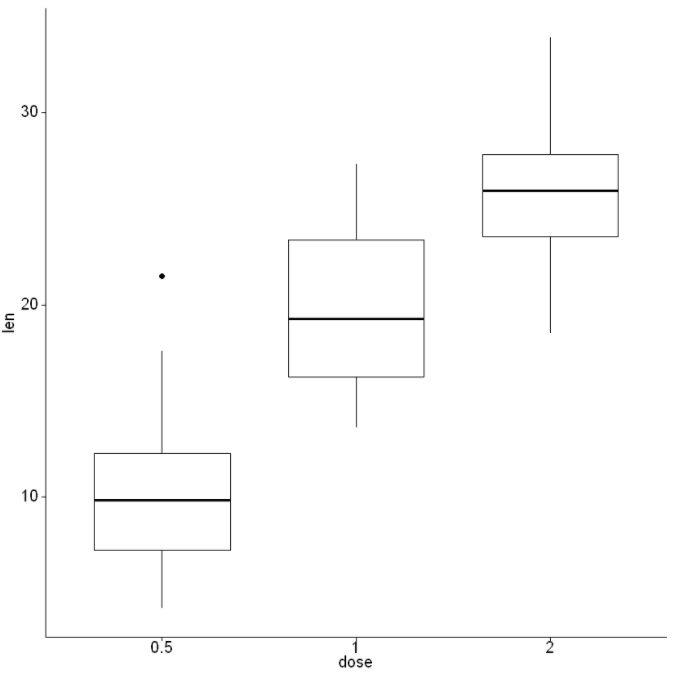
2. 更改绘图方向
可选参数:"vertical", "horizontal", "reverse" (“垂直”,“水平”,“反向”)
In[4]:
ggpar(p, orientation = "vertical")Out[4]:
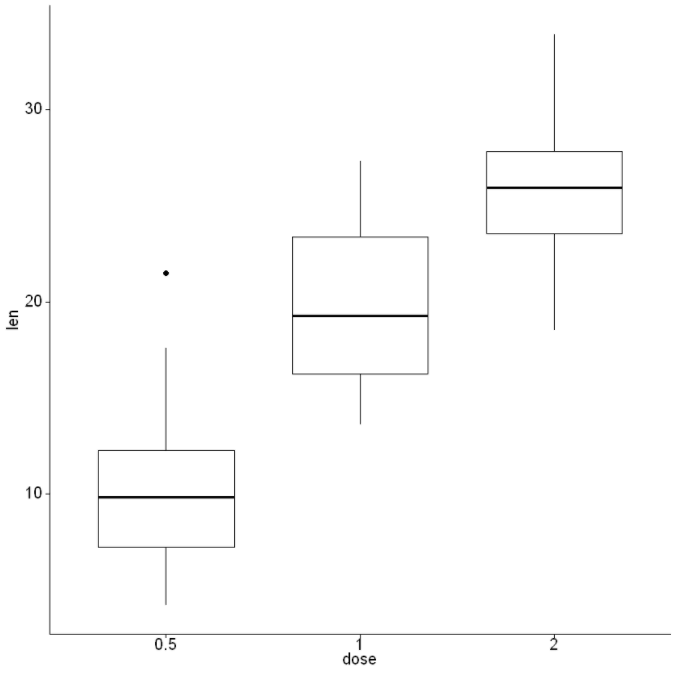
In[5]:
ggpar(p, orientation = "reverse")Out[5]:
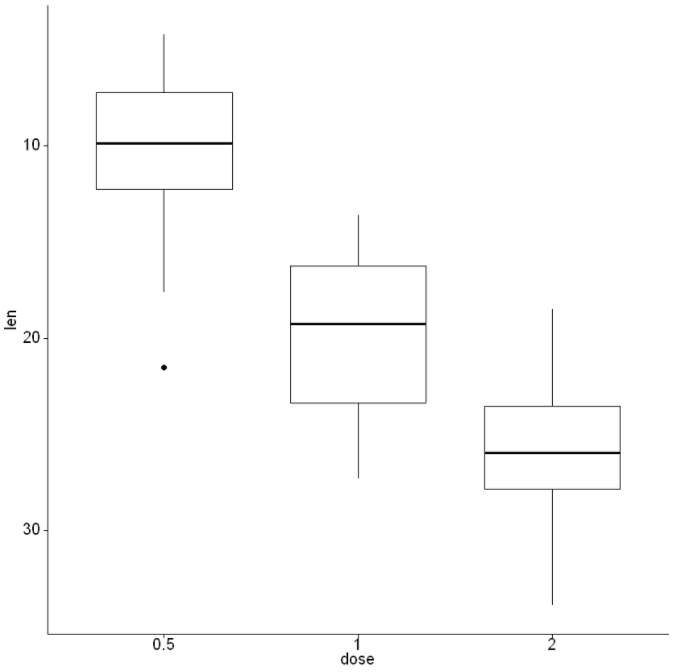
In[6]:
ggpar(p, orientation = "horizontal")Out[6]:
rotate 如果为TRUE,则通过将绘图方向设置为水平来旋转图形。
In[7]:
ggpar(p,rotate = T)Out[7]:

1. 更改标题名和轴标签名
In[8]:
ggpar(p, main = "Plot of length n by dose", xlab = "Dose (mg)", ylab = "Length")Out[8]:
2. 更改标题和轴标签样式
标题字体样式: 'plain', 'italic', 'bold', 'bold.italic'(“普通”,“斜体”,“粗体”,“斜粗体”)
In[9]:
ggpar(p, main = "Length by dose", submain = "plot by length", font.main = c(24,"bold.italic", "red"), font.submain = c(14,"bold.italic", "green"), font.x = c(14, "bold", "#2E9FDF"), font.y = c(14, "bold", "#E7B800"))Out[9]:
3. 隐藏轴标签
In[10]:
ggpar(p, xlab = FALSE, ylab = FALSE)Out[10]:

1. 带颜色的箱体图
In[11]:
p2 <- ggboxplot(df, "dose", "len", color = "dose") p2Out[11]:
2. 使用自定义调色板
In[12]:
ggpar(p2, palette = c("#00AFBB", "#E7B800", "#FC4E07"))
Out[12]:
3. 使用Dark2 、grey 、 npg等调色板
In[13]:
ggpar(p2, palette = "Dark2" )Out[13]:
In[14]:
ggpar(p2, palette = "grey")Out[14]:
In[15]:
ggpar(p2, palette = "npg") # natureOut[15]:

1. 轴刻度标签和旋转
font.tickslab, font.xtickslab, font.ytickslab:刻度标签的字体样式(大小,字体,颜色)
x.text.angle, y.text.angle:x、y轴的字体方向
In[16]:
ggpar(p, font.tickslab = c(14,"bold", "#993333"), x.text.angle = 45, y.text.angle = 45)Out[16]:
2. 隐藏轴刻度和刻度标签
In[17]:
ggpar(p, ticks = F, tickslab = F)Out[17]:
3. 设置轴的范围
In[18]:
ggpar(p, ylim = c(-20, 60))Out[18]:
In[19]:
ggpar(p, ylim = c(-20, 60),yticks.by = 10 )Out[19]:
4. 轴刻度log2转换
In[20]:
ggpar(p, yscale = "log2", format.scale = TRUE)Out[20]:

1. 更改图例的位置和标题
In[21]:
ggpar(p2, legend = "right", legend.title = "Dose (mg)", font.legend = c(10, "bold", "red"))Out[21]:

往期相关链接:
1、R基础篇
excel不熟练怎么办,R来帮您(一)数据分类汇总; R相关软件及R包安装; 【零基础学绘图】之绘制venn图(五);2、R进阶
【绘图进阶】之六种带中心点的PCA 图和三维PCA图绘制(四);
【绘图进阶】之交互式可删减分组和显示样品名的PCA 图(三);
3.python基础篇
4、数据提交
3分钟学会CHIP-seq类实验测序数据可视化 —IGV的使用手册;
10分钟搞定多样性数据提交,最快半天内获取登录号,史上最全的多样性原始数据提交教程;
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
5、表达谱分析
表达谱分析(二)通路富集分析和基因互作网络图绘制;6、医学数据分析
KING: 样本亲缘关系鉴定工具;【WGS服务升级】人工智能软件SpliceAI助力解读罕见和未确诊疾病中的非编码突变;
隐性疾病trio家系别忽视单亲二倍体现象——天昊数据分析助力临床疾病诊断新添UPD(单亲二倍体)可视化分析工具;
【昊工具】Oh My God! 太好用了吧!疾病或表型的关键基因查询数据库,我不允许你不知道Phenolyzer;
天昊客户服务中心
手机/微信号:18964693703

【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849

作者:大熊
审核:有才
来源:天昊生信团